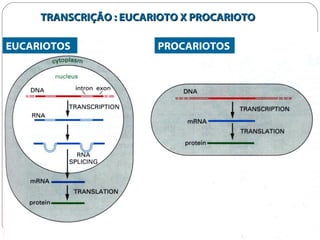
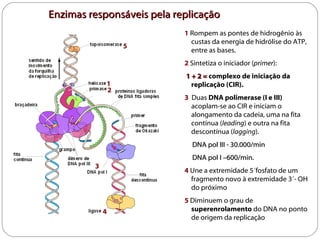
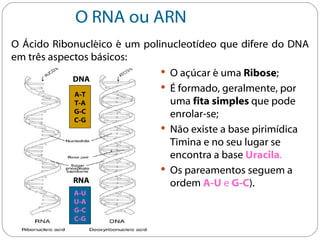
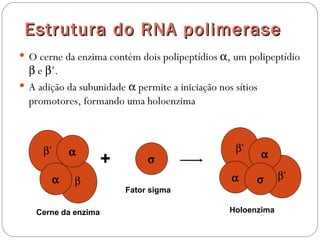
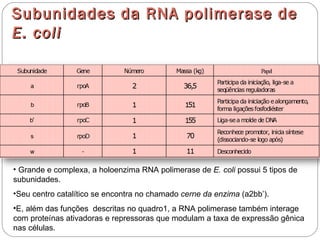
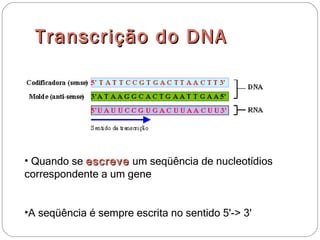
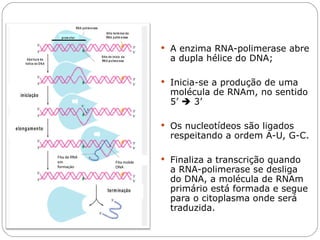
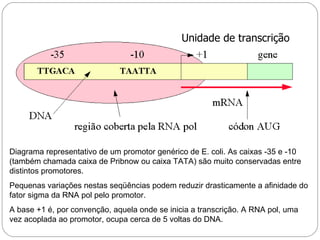
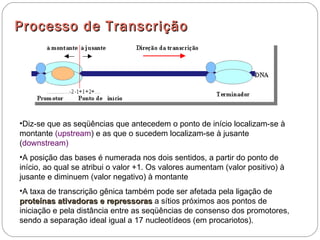
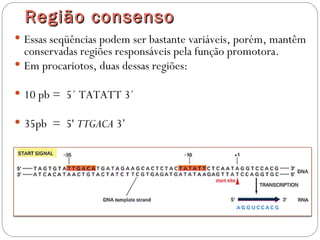
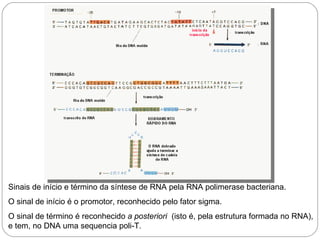
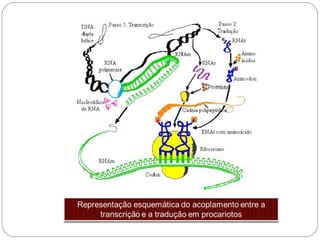
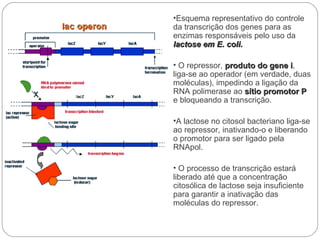
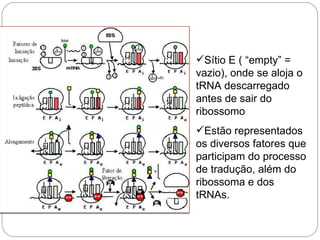
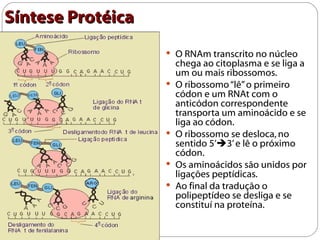
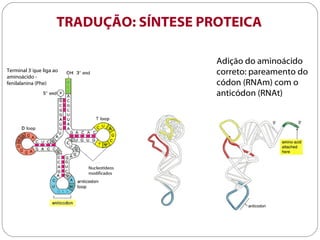
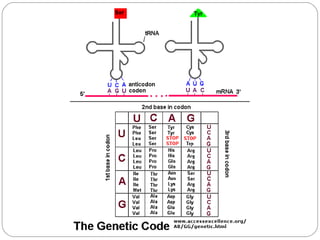
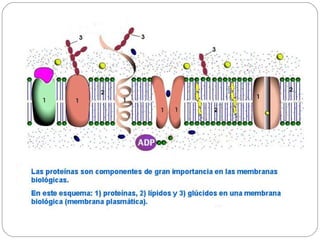
O documento descreve os processos de transcrição e regulação gênica em bactérias. A transcrição envolve as etapas de iniciação, alongamento e término, mediadas pela RNA polimerase e reconhecimento de seqüências específicas de DNA como promotores e terminadores. A expressão gênica pode ser regulada em diferentes níveis como transcrição, processamento e tradução do RNA.