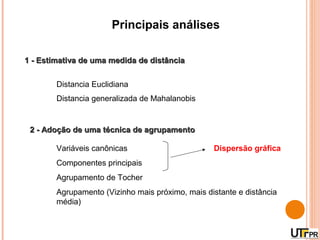
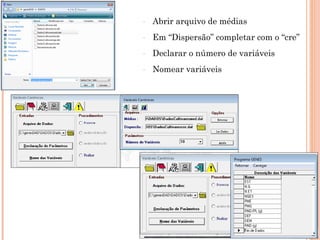
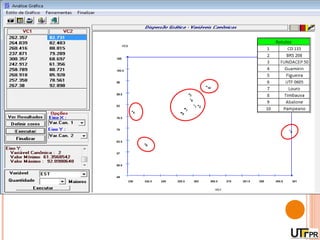
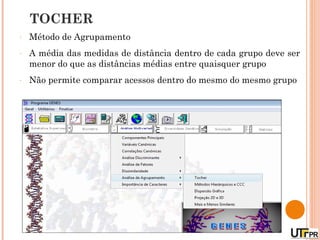
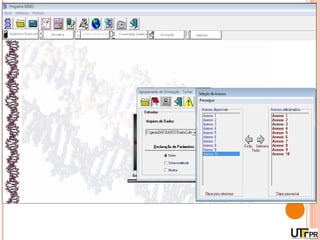
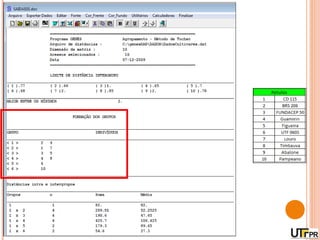
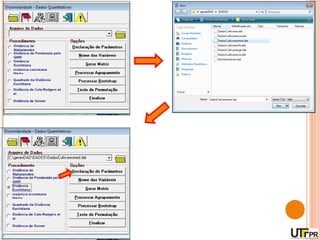
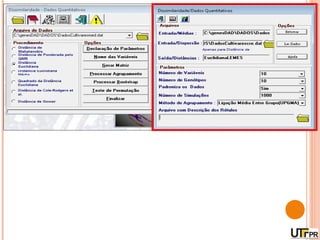
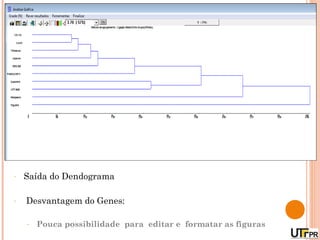
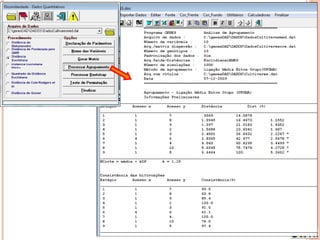
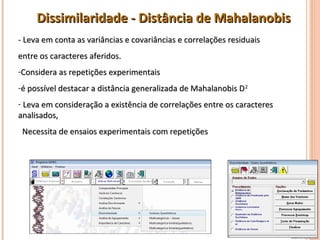
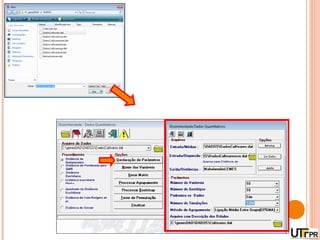
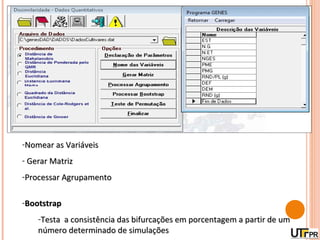
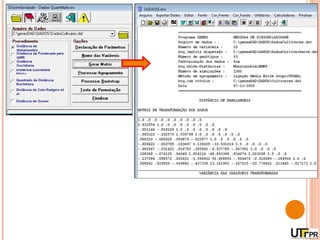
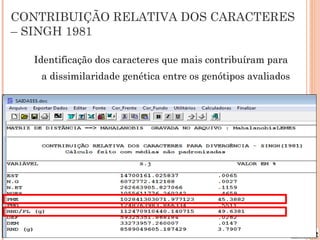
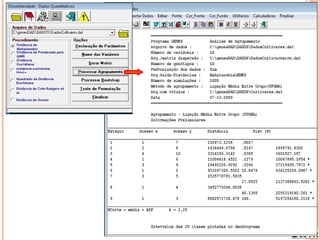
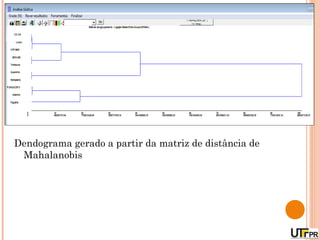
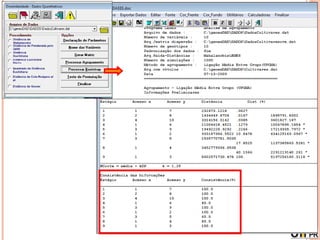
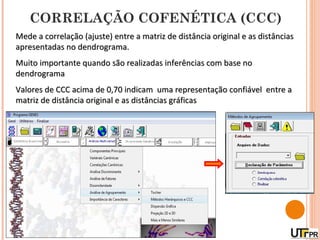
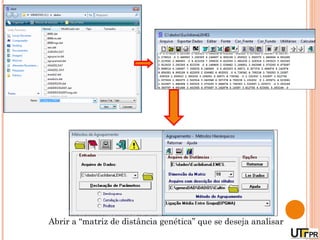
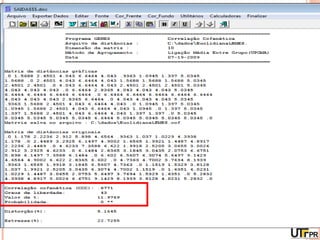
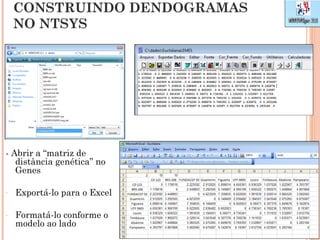
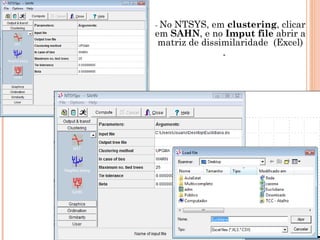
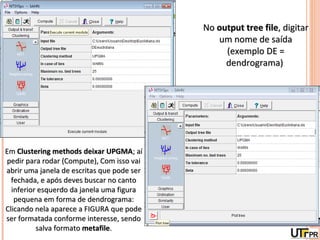
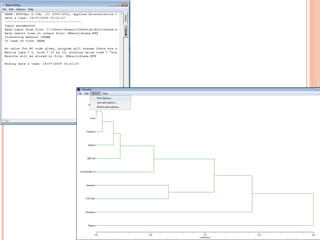
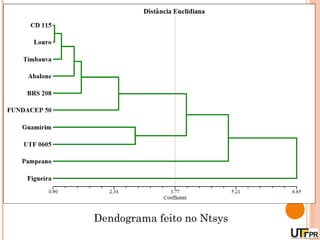
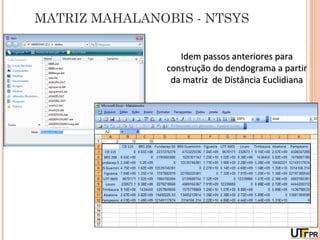
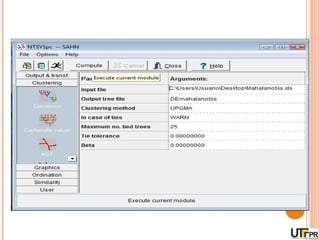
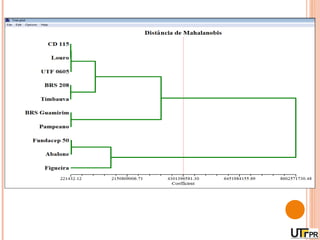
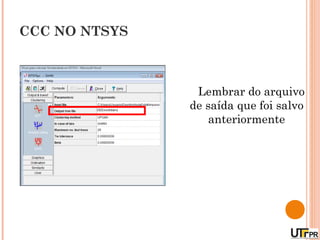
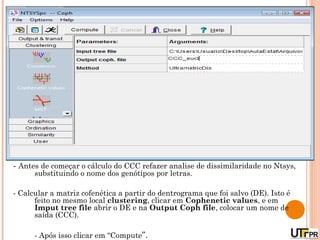
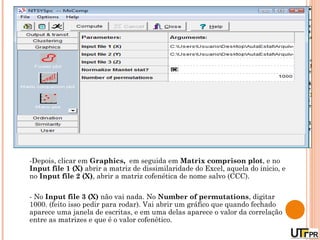
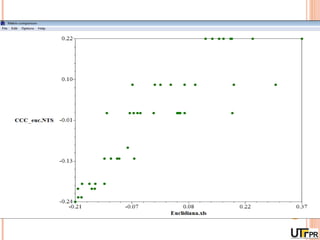
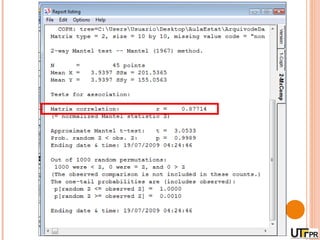
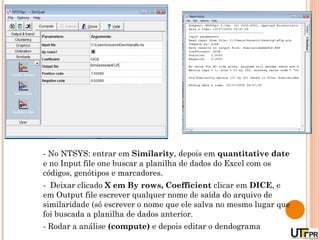
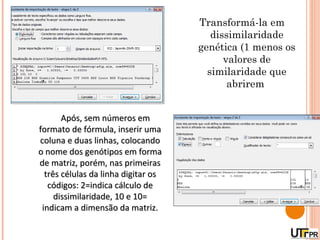
Este documento discute técnicas de agrupamento e dissimilaridade para análises genéticas. Aborda métodos como variáveis canônicas, componentes principais e distância de Mahalanobis. Também explica como construir dendogramas no programa NTSYS e calcular a correlação cofenética.