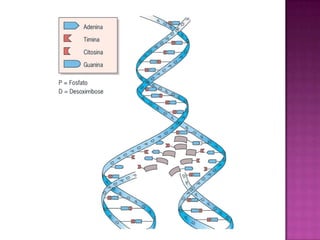
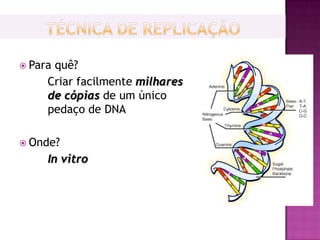
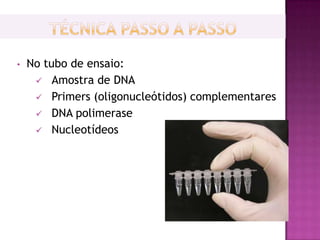
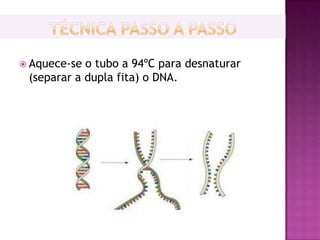
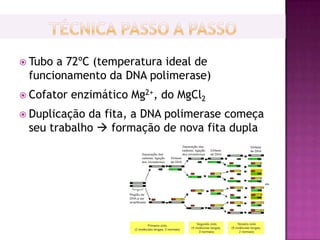
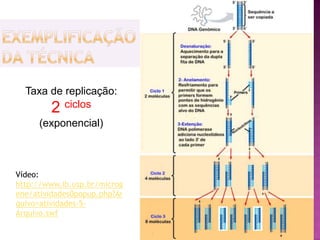
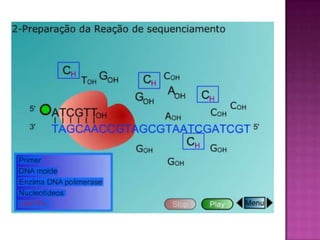
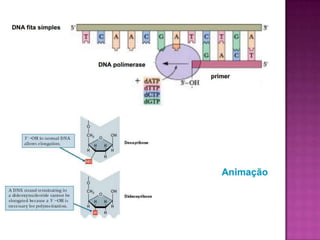
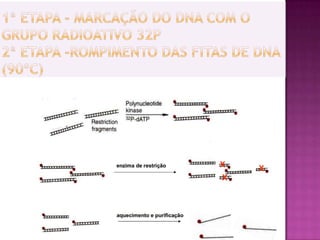
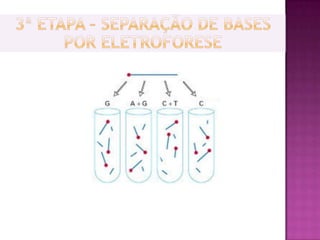
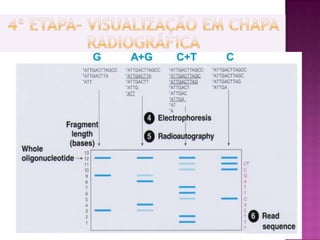
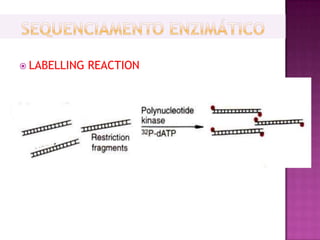
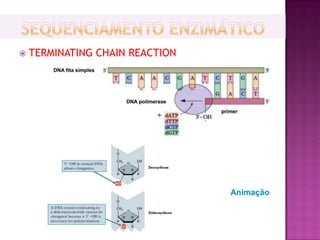
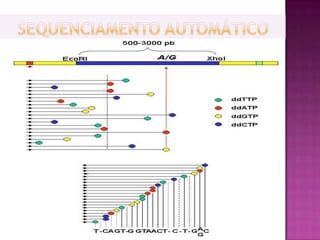
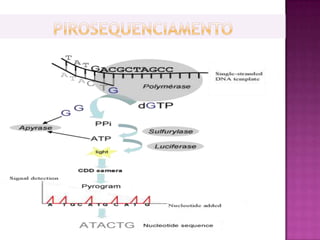
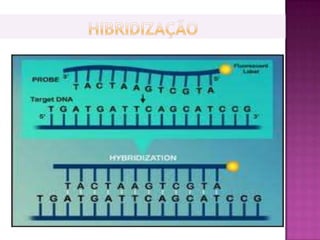
O documento descreve o processo de Reação em Cadeia da Polimerase (PCR), que permite amplificar seletivamente fragmentos de DNA. A PCR usa primers, nucleotídeos e a enzima DNA polimerase para replicar um pedaço específico de DNA in vitro, gerando milhares de cópias do fragmento. O processo consiste em ciclos de aquecimento, arrefecimento e extensão para replicar o DNA alvo.