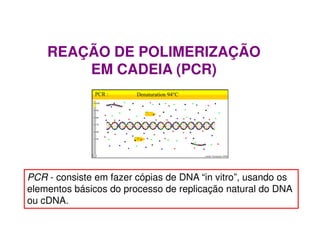
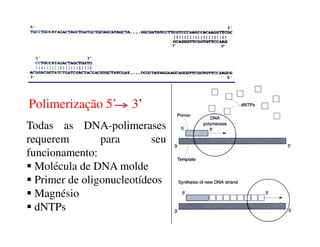
A Reação de Polimerização em Cadeia (PCR) é uma técnica que permite a amplificação de DNA in vitro, utilizando polimerase termoestável e seguindo três etapas: desnaturação, anelamento e extensão. Desenvolvida por Kary Mullis nos anos 1980, essa técnica envolve ciclos de temperatura que facilitam a separação e replicação das fitas de DNA, com o uso de primers específicos. A eficiência da PCR pode variar de 78% a 97% devido a fatores como a presença de contaminantes e a qualidade dos reagentes.