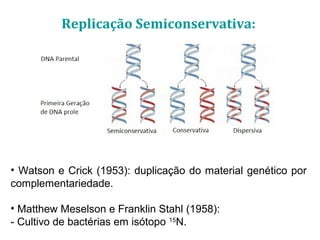
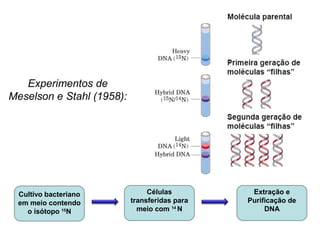
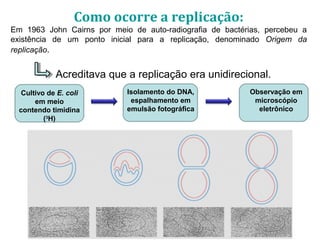
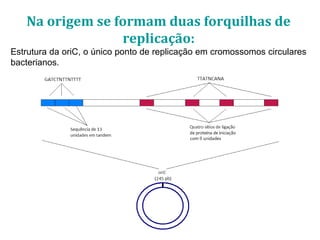
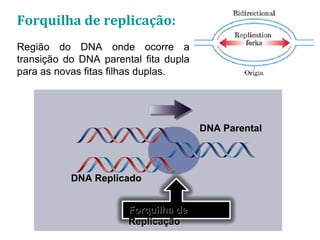
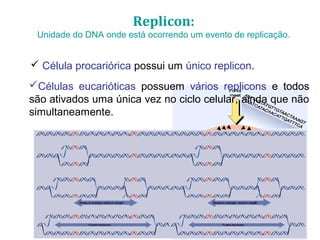
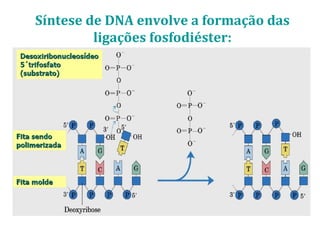
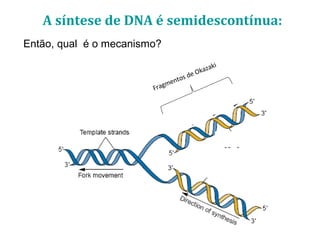
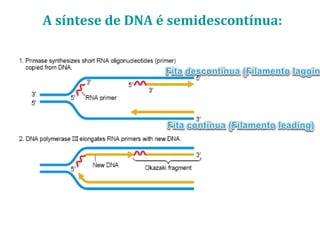
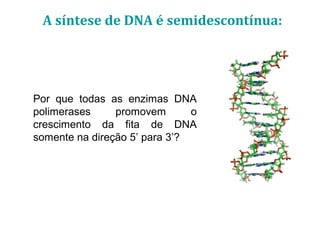
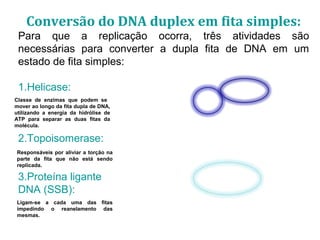
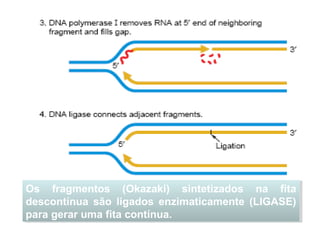
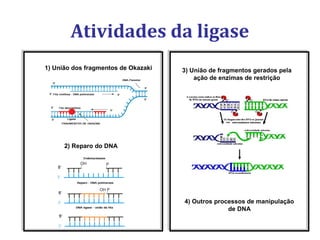
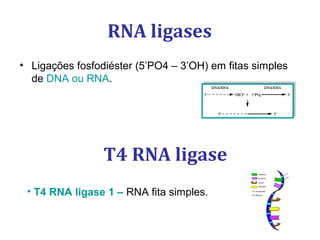
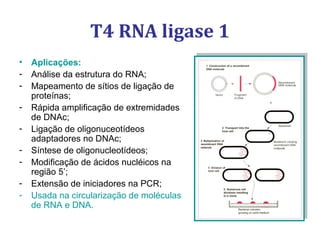
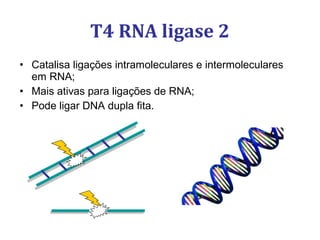
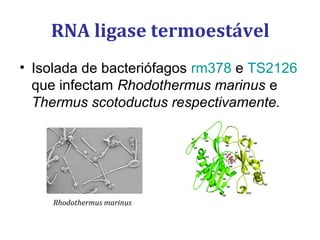
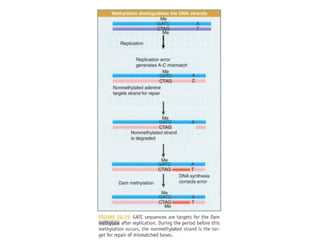
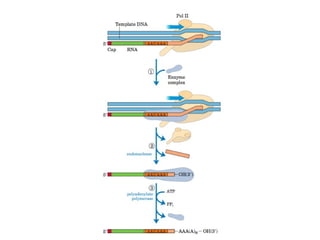
[1] O documento descreve os principais processos da replicação do DNA em bactérias, incluindo a replicação semiconservativa, os fragmentos de Okazaki, e as enzimas envolvidas como DNA polimerases e helicases.
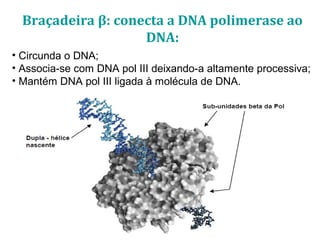
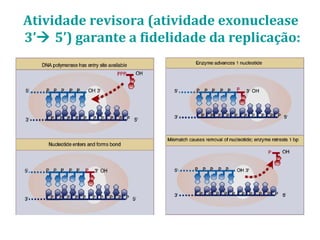
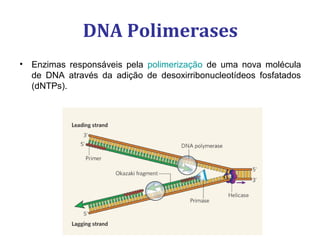
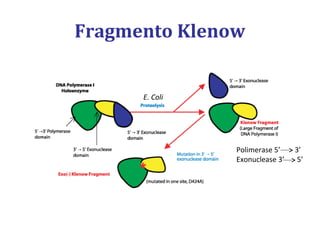
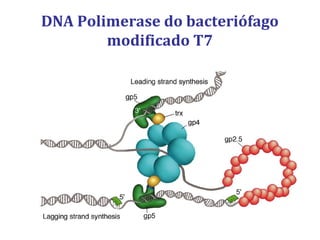
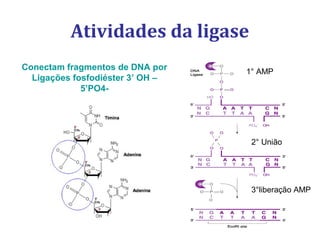
[2] Apresenta detalhes sobre as DNA polimerases bacterianas, como a Pol I, Pol II e Pol III, e suas funções na replicação e reparo do DNA.
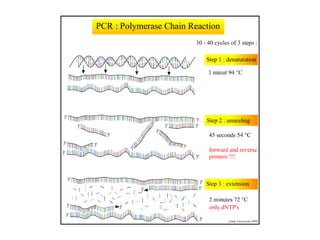
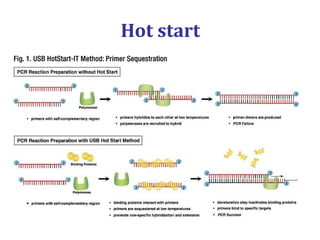
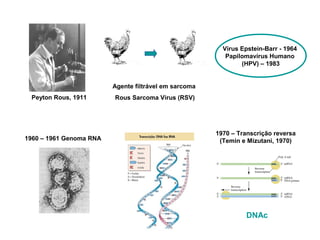
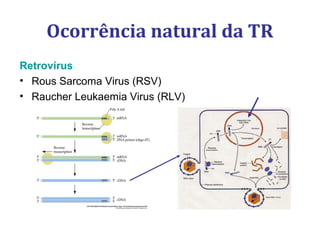
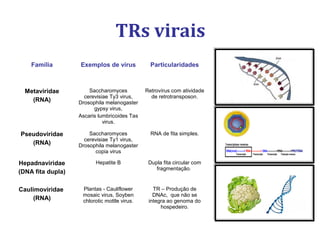
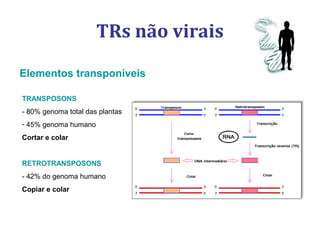
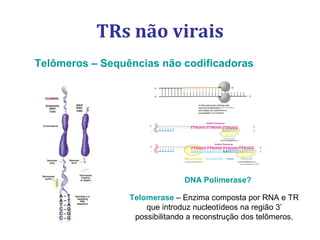
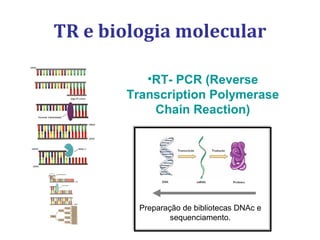
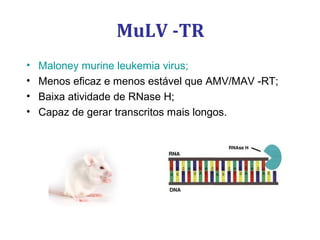
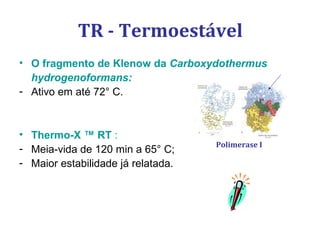
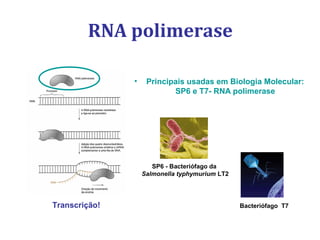
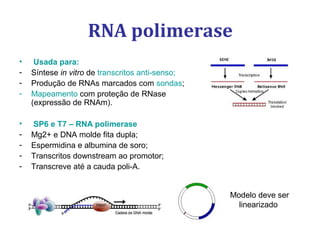
[3] Discutem transcriptases reversas virais e não virais, e seu papel na biologia molecular, como a RT-PCR.