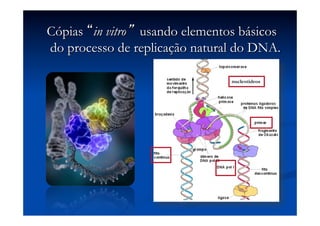
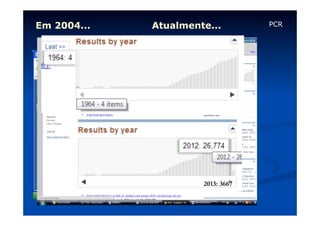
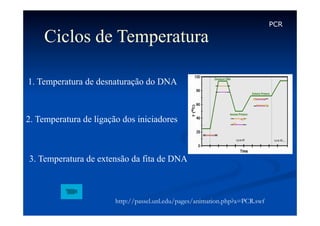
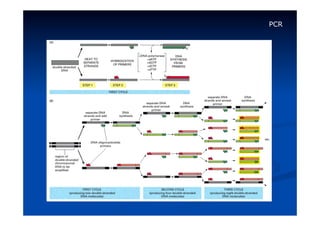
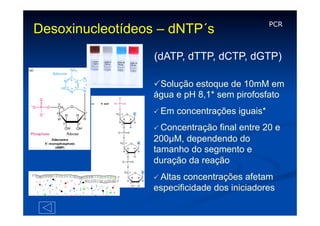
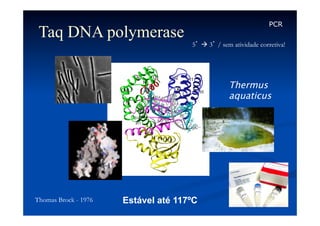
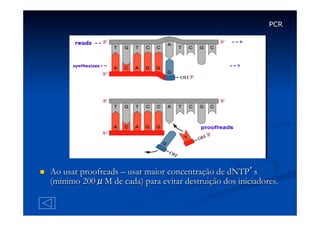
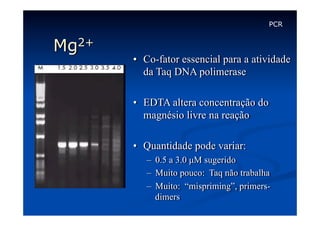
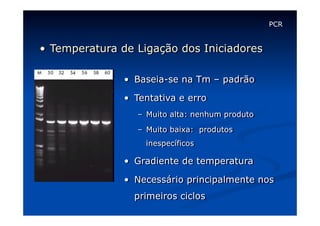
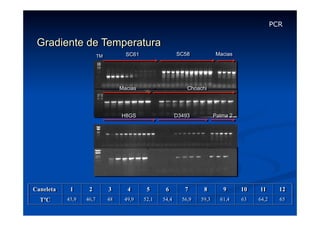
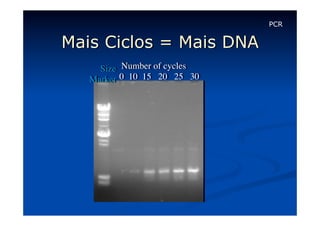
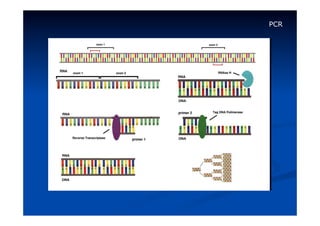
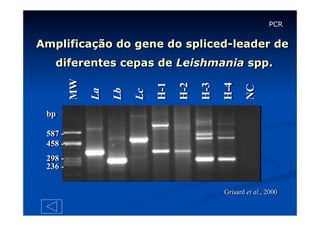
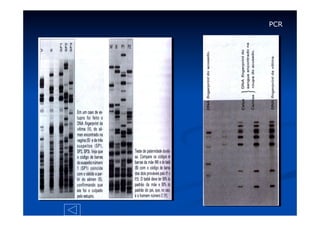
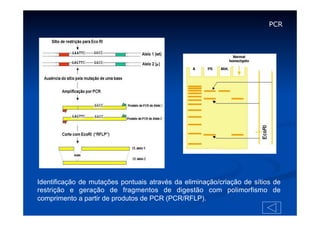
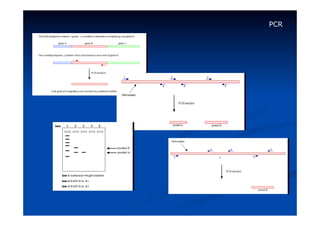
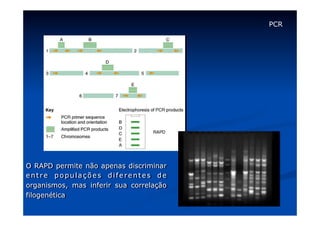
Este documento descreve a reação em cadeia da polimerase (PCR), incluindo seus reagentes, etapas e aplicações. A PCR permite a amplificação seletiva de segmentos específicos de DNA e foi inventada por Kary Mullis em 1983, revolucionando a biologia molecular.