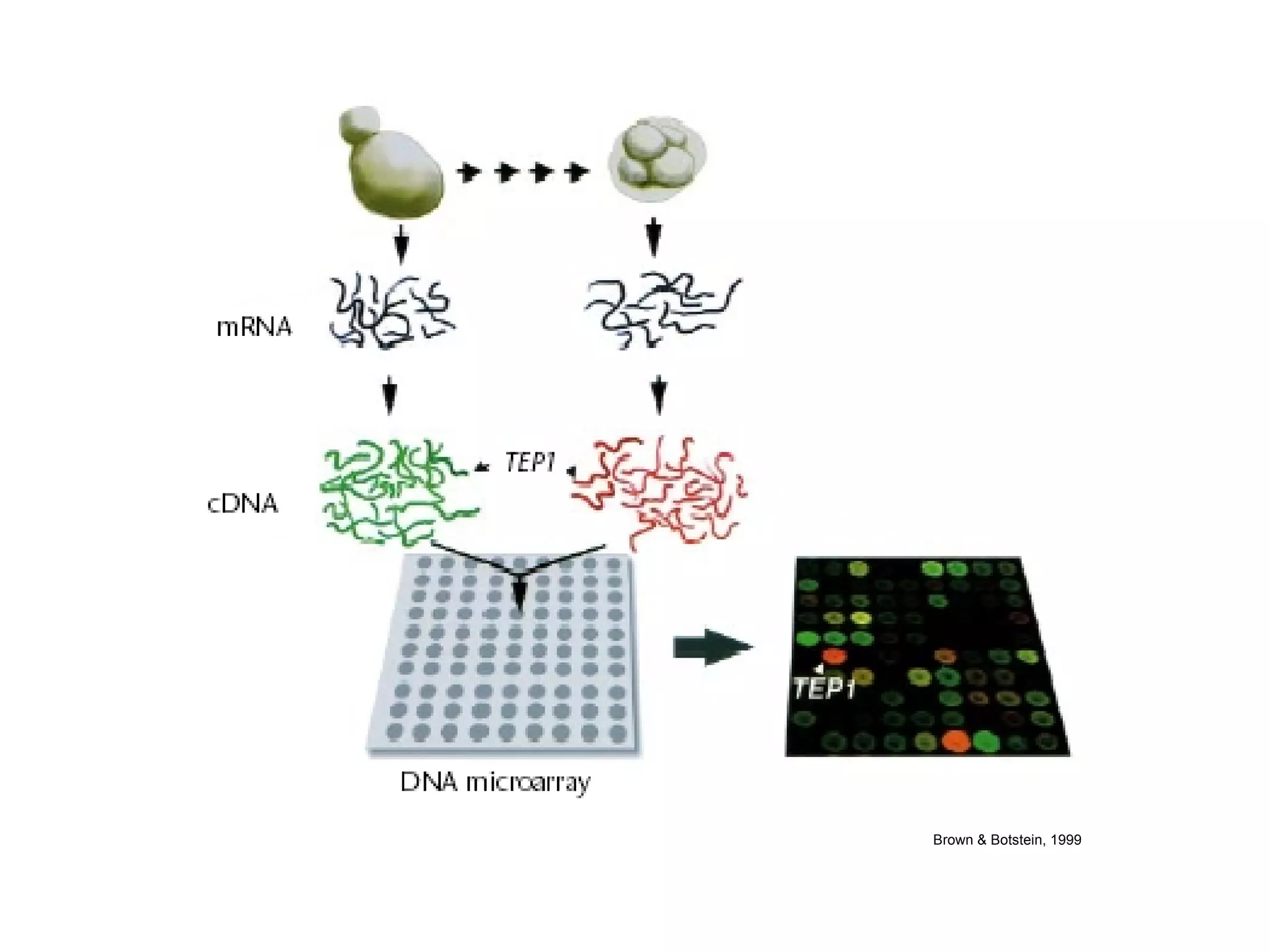
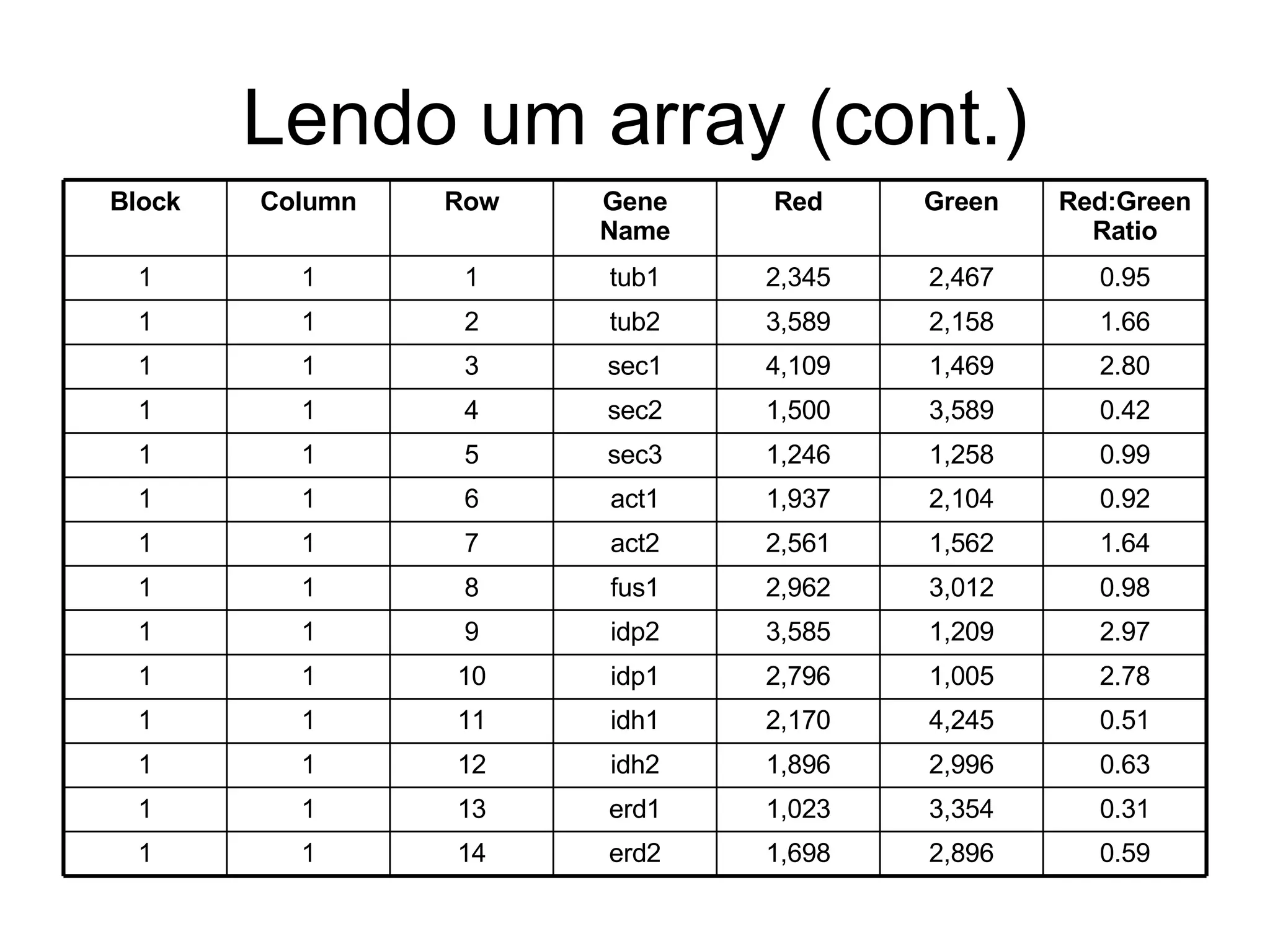
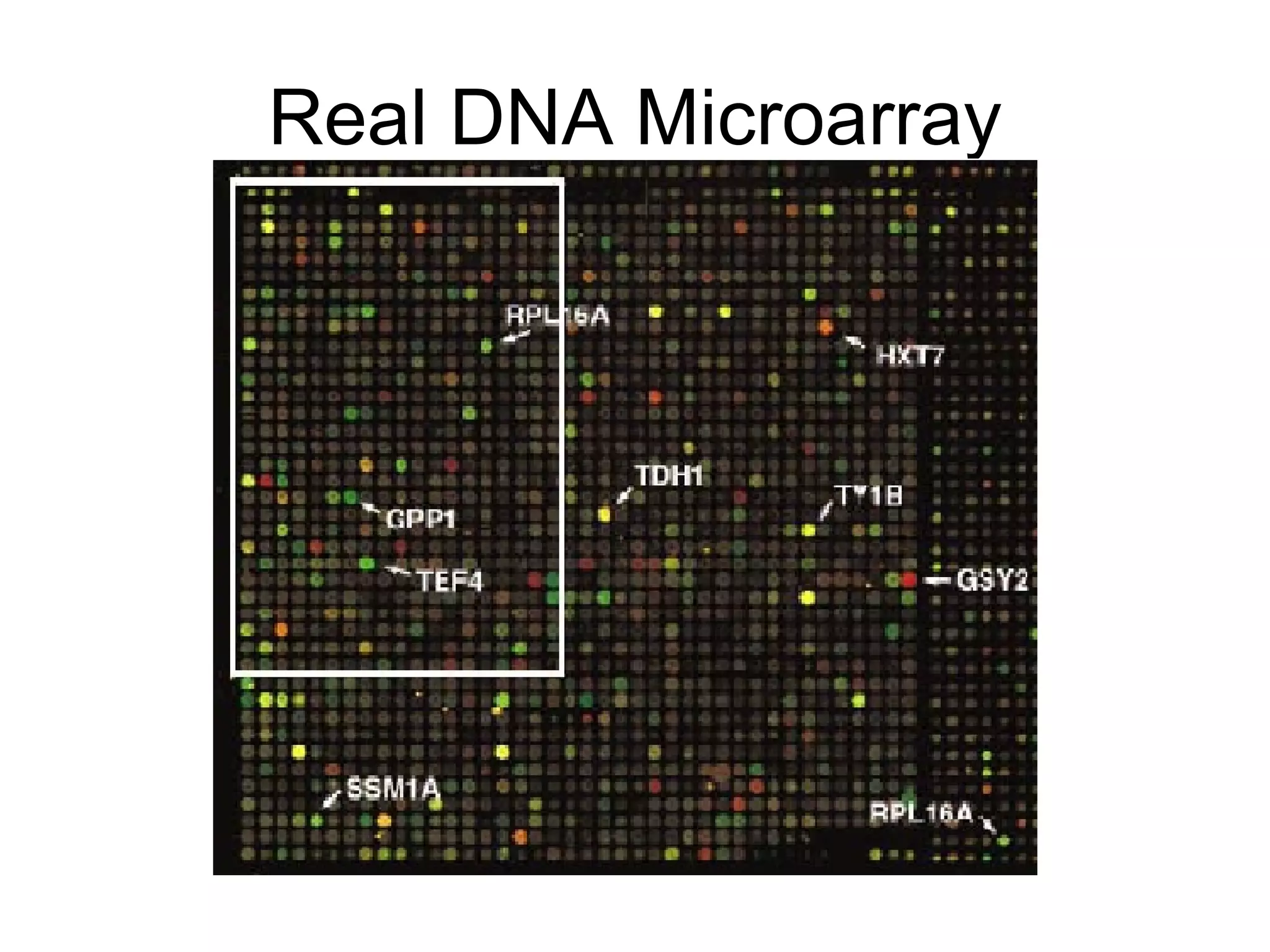
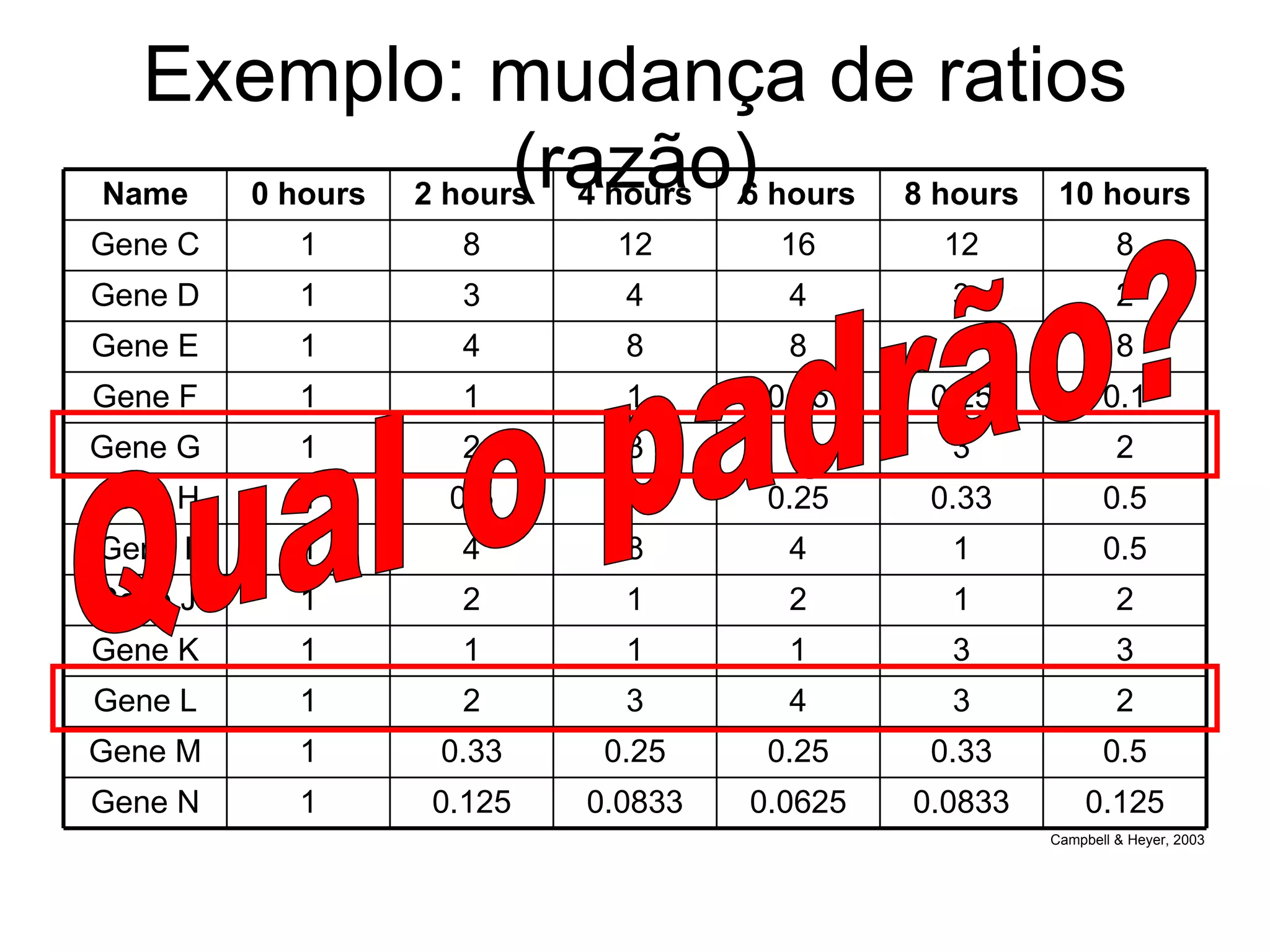
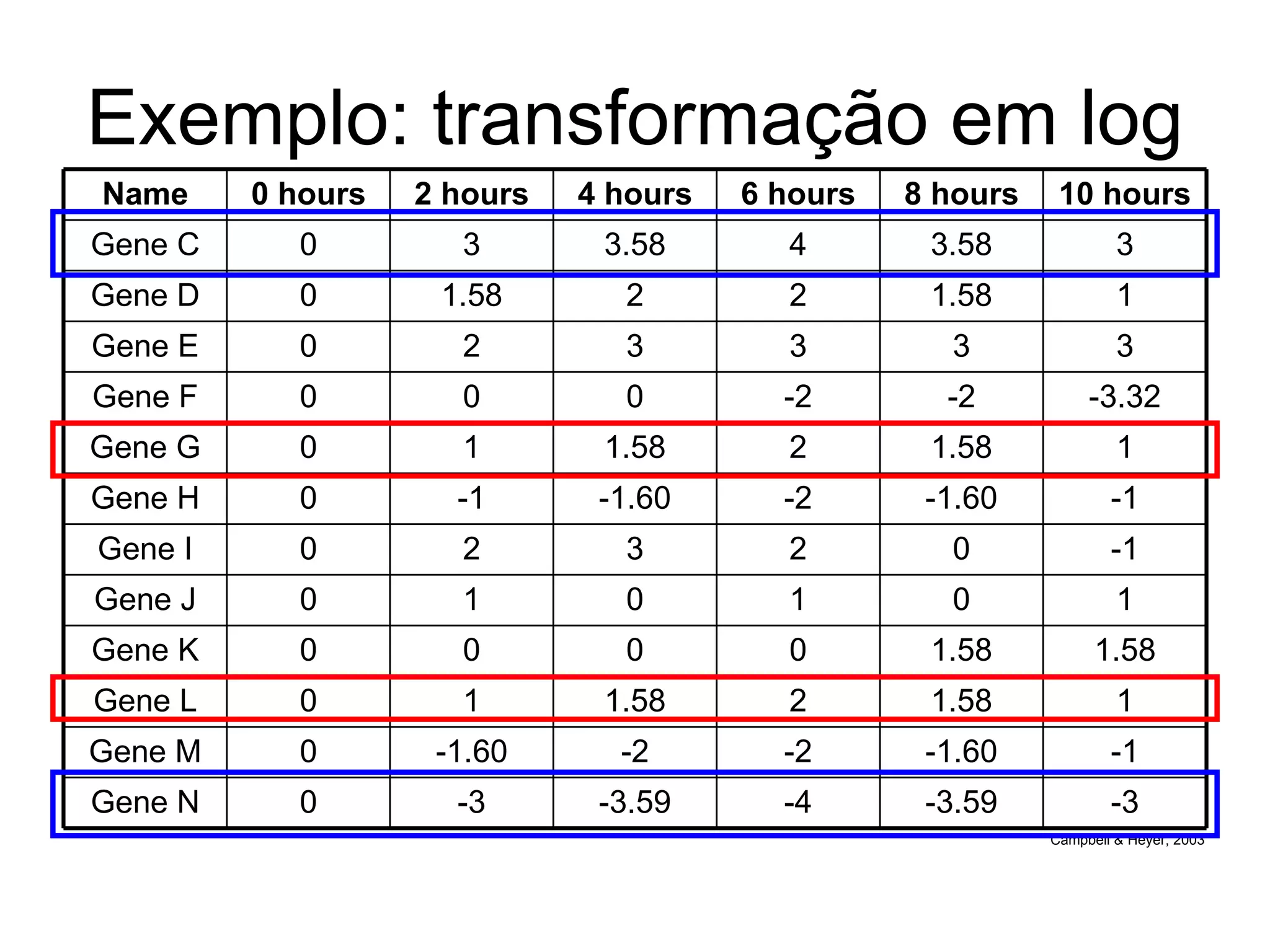
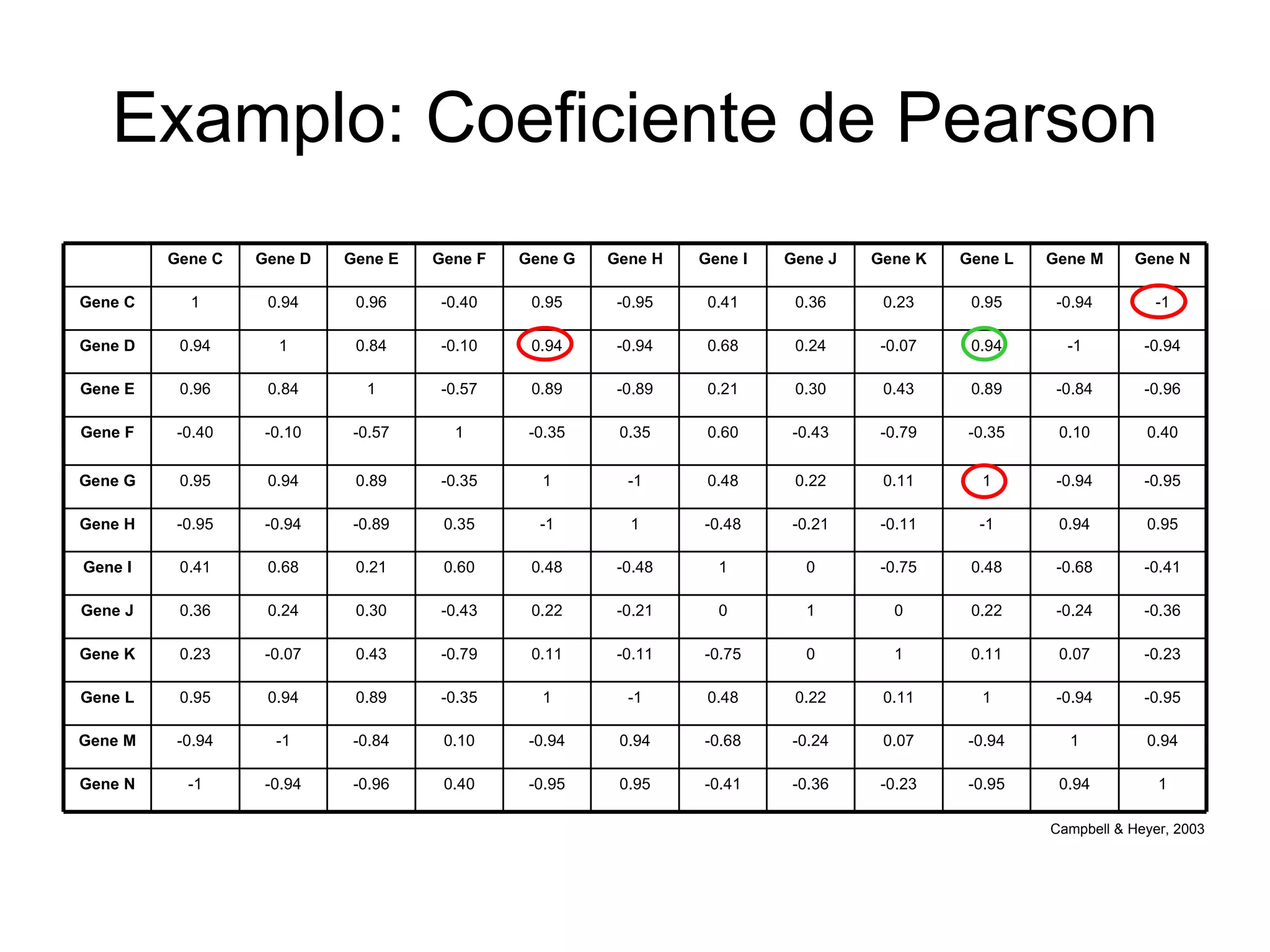
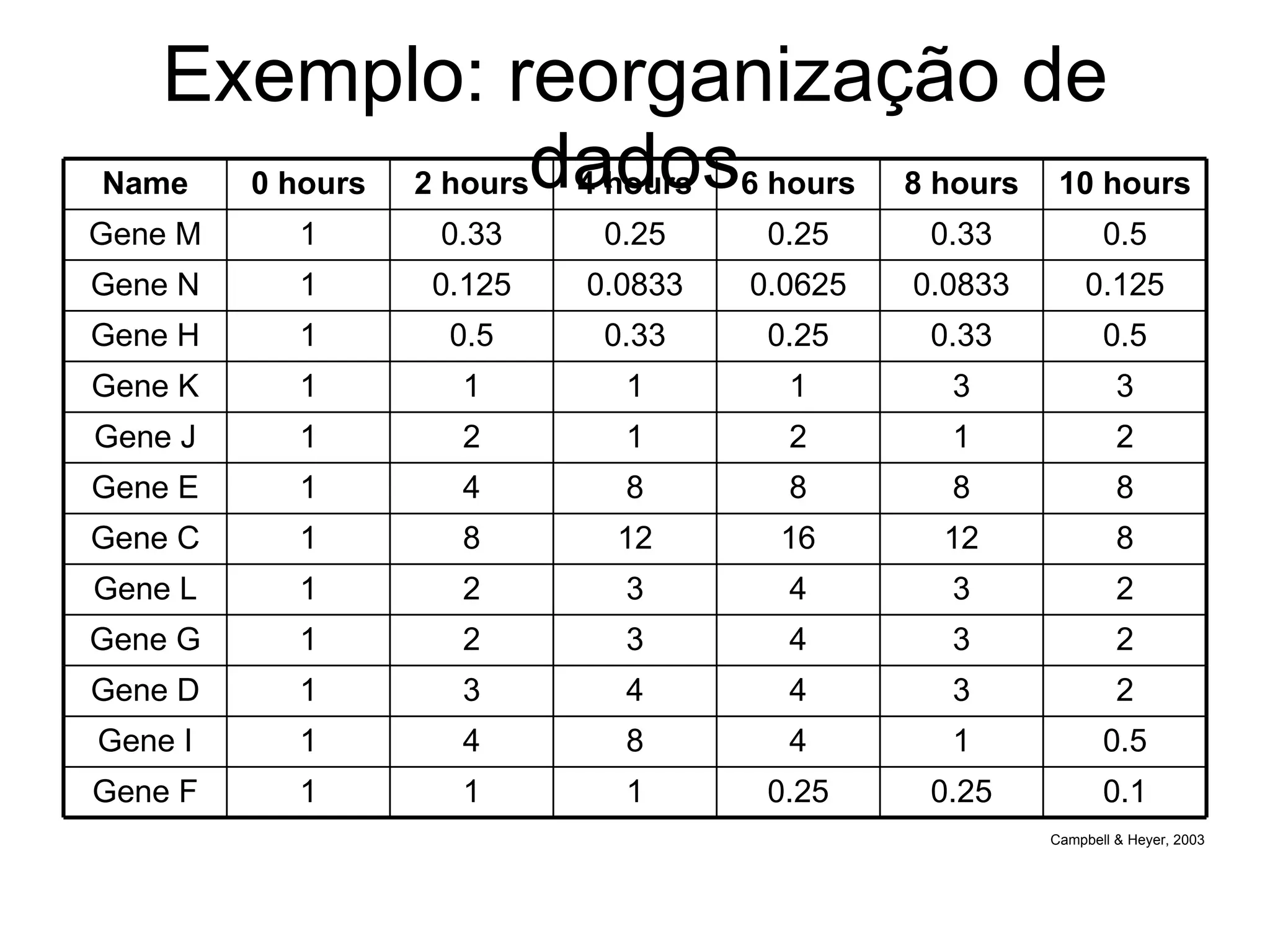
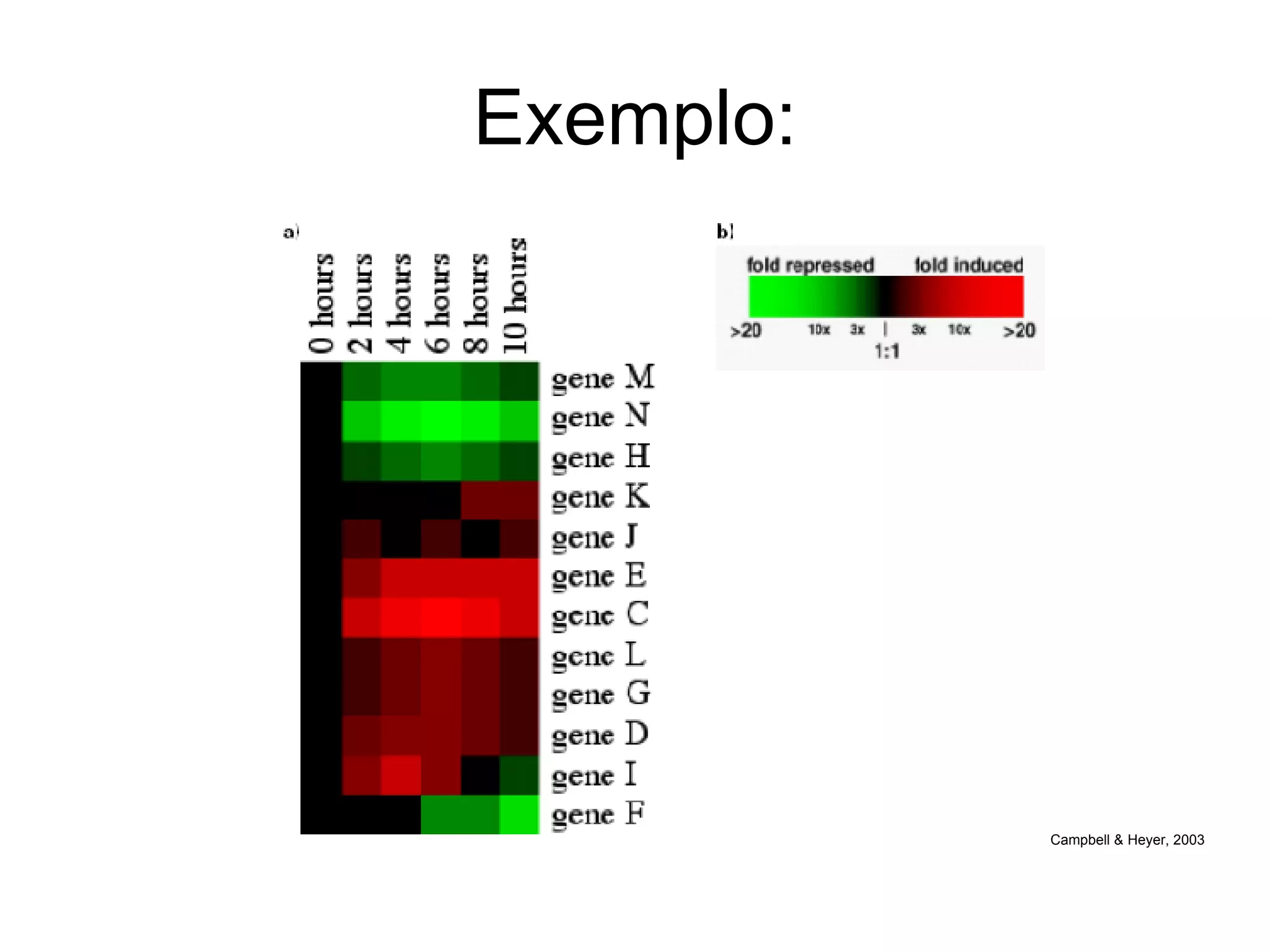
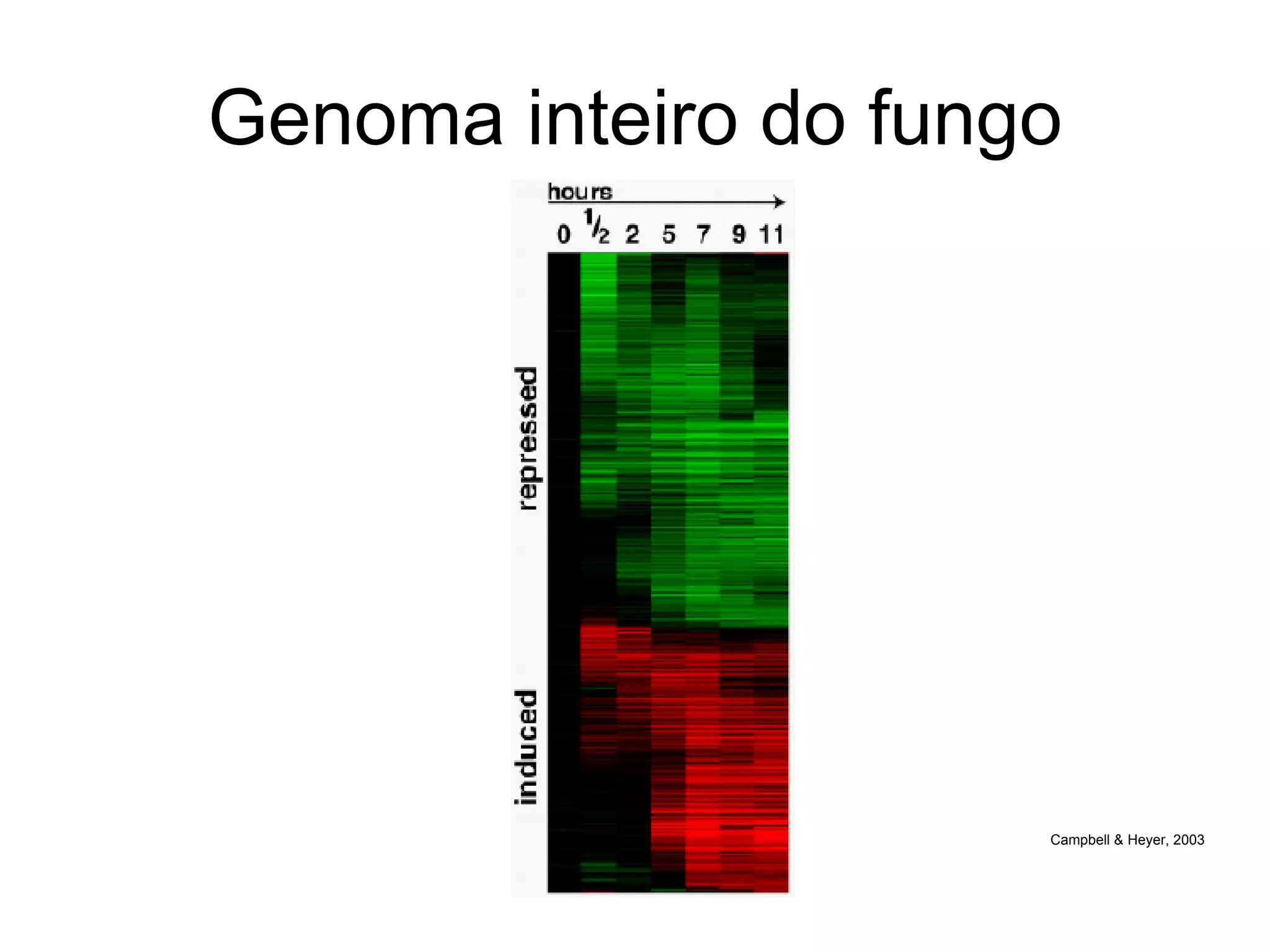
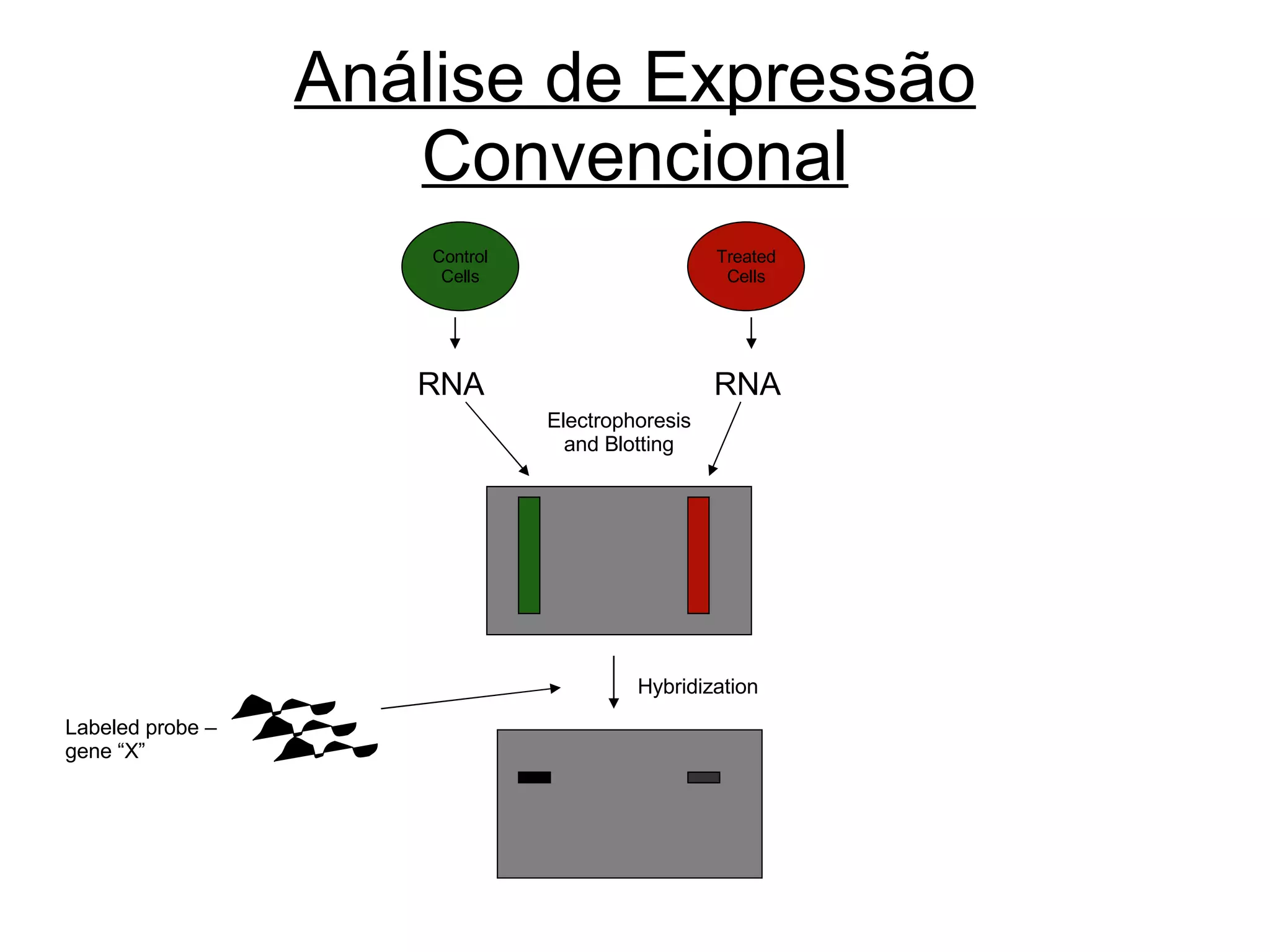
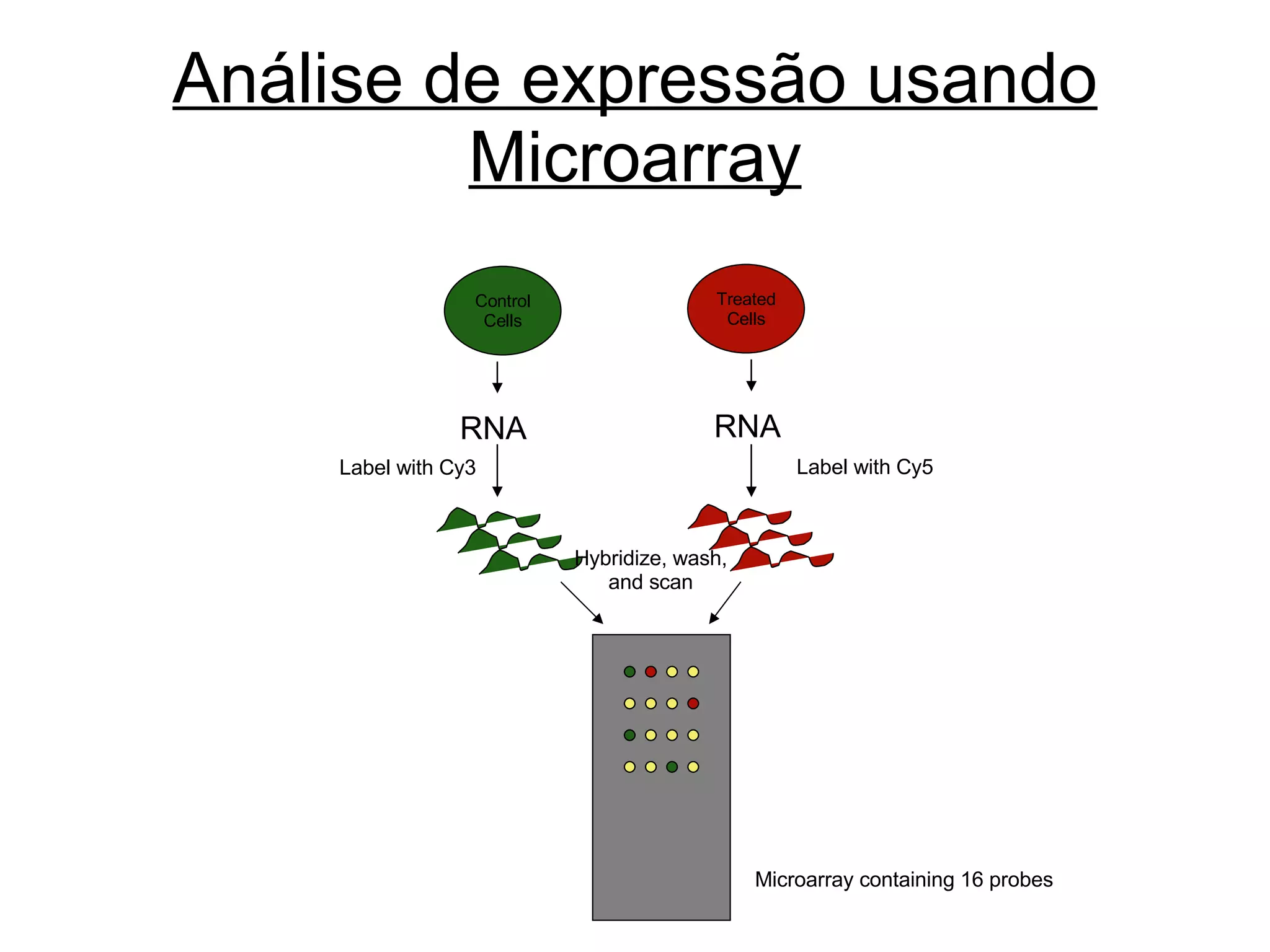
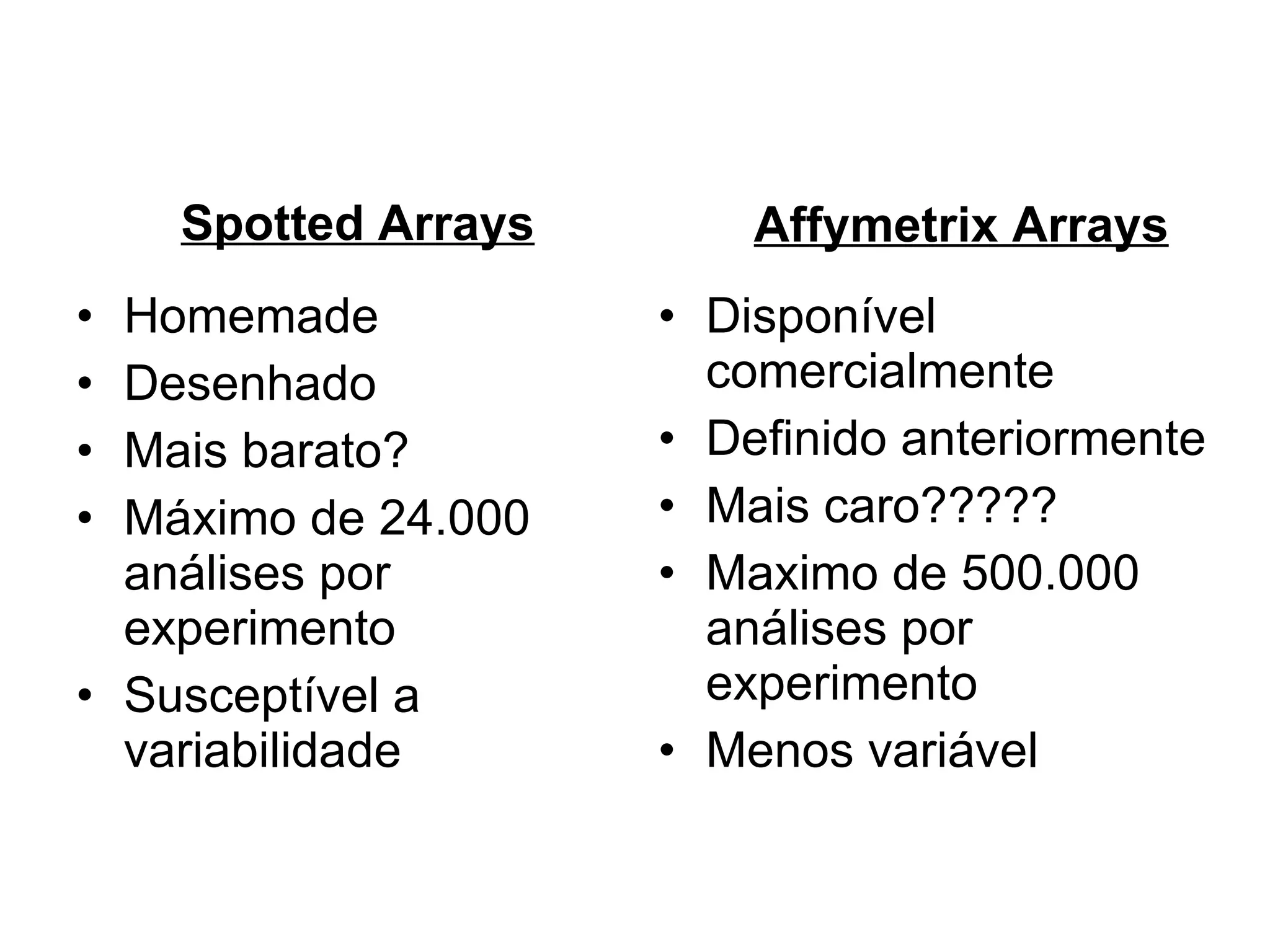
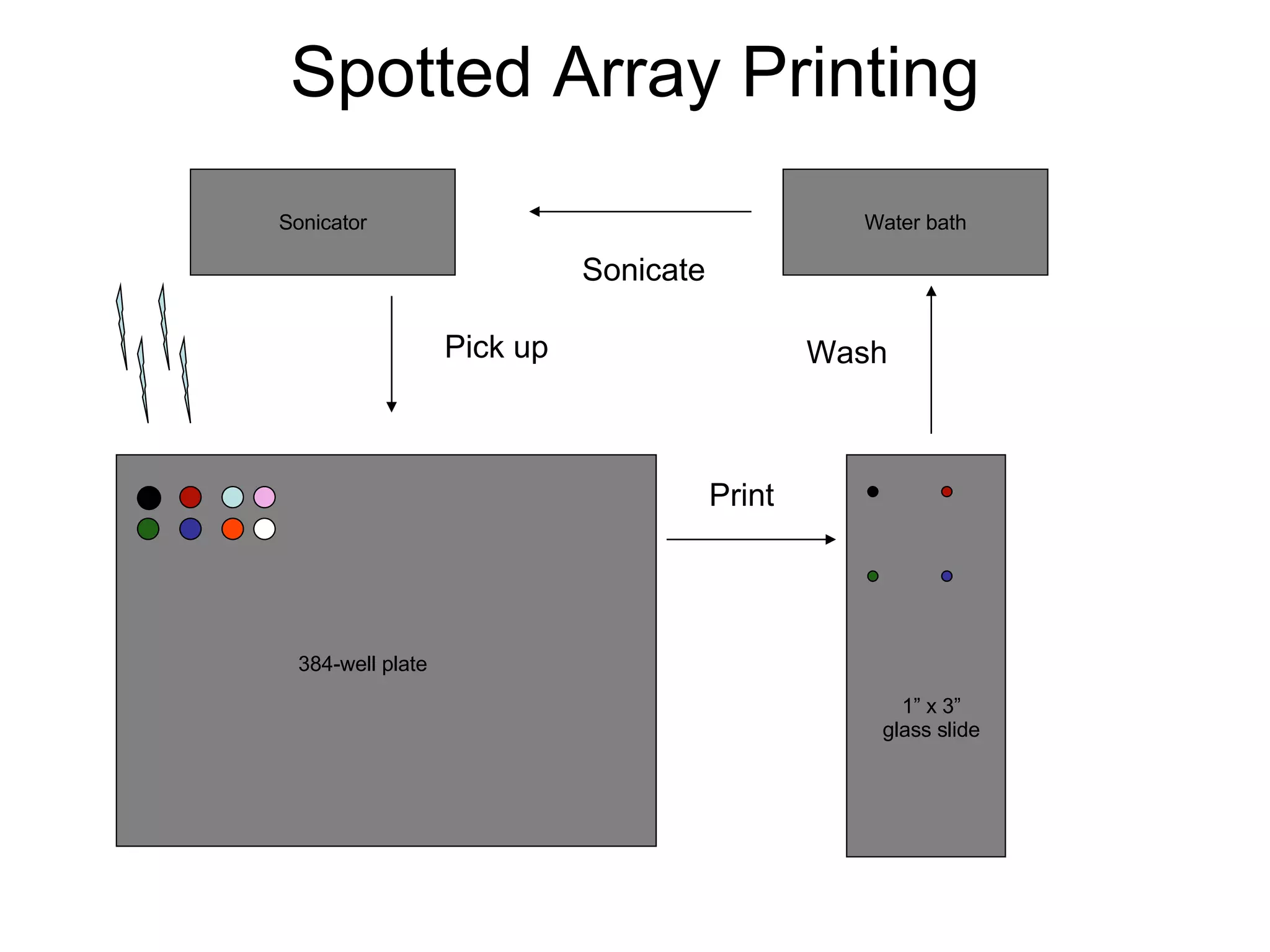
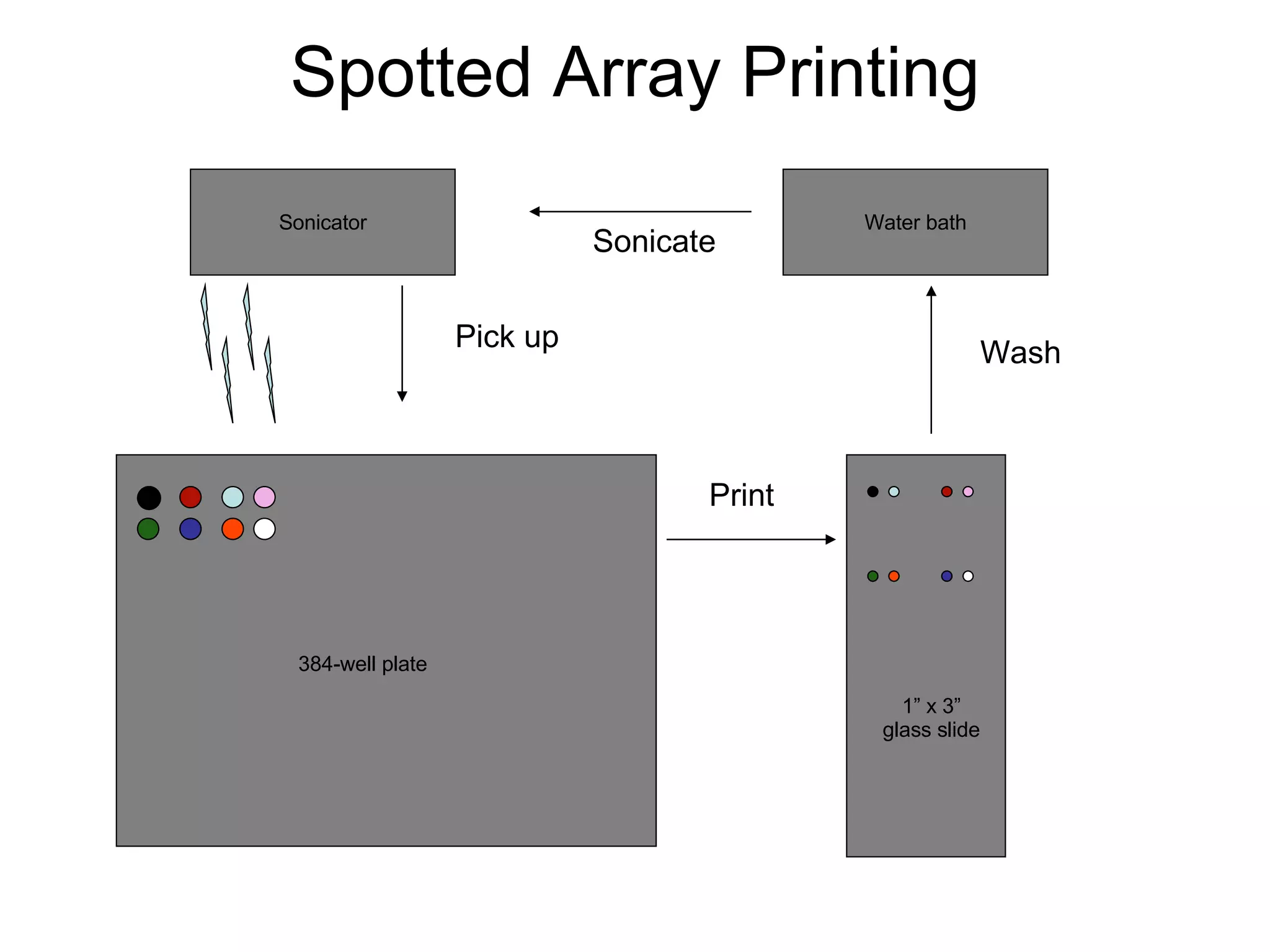
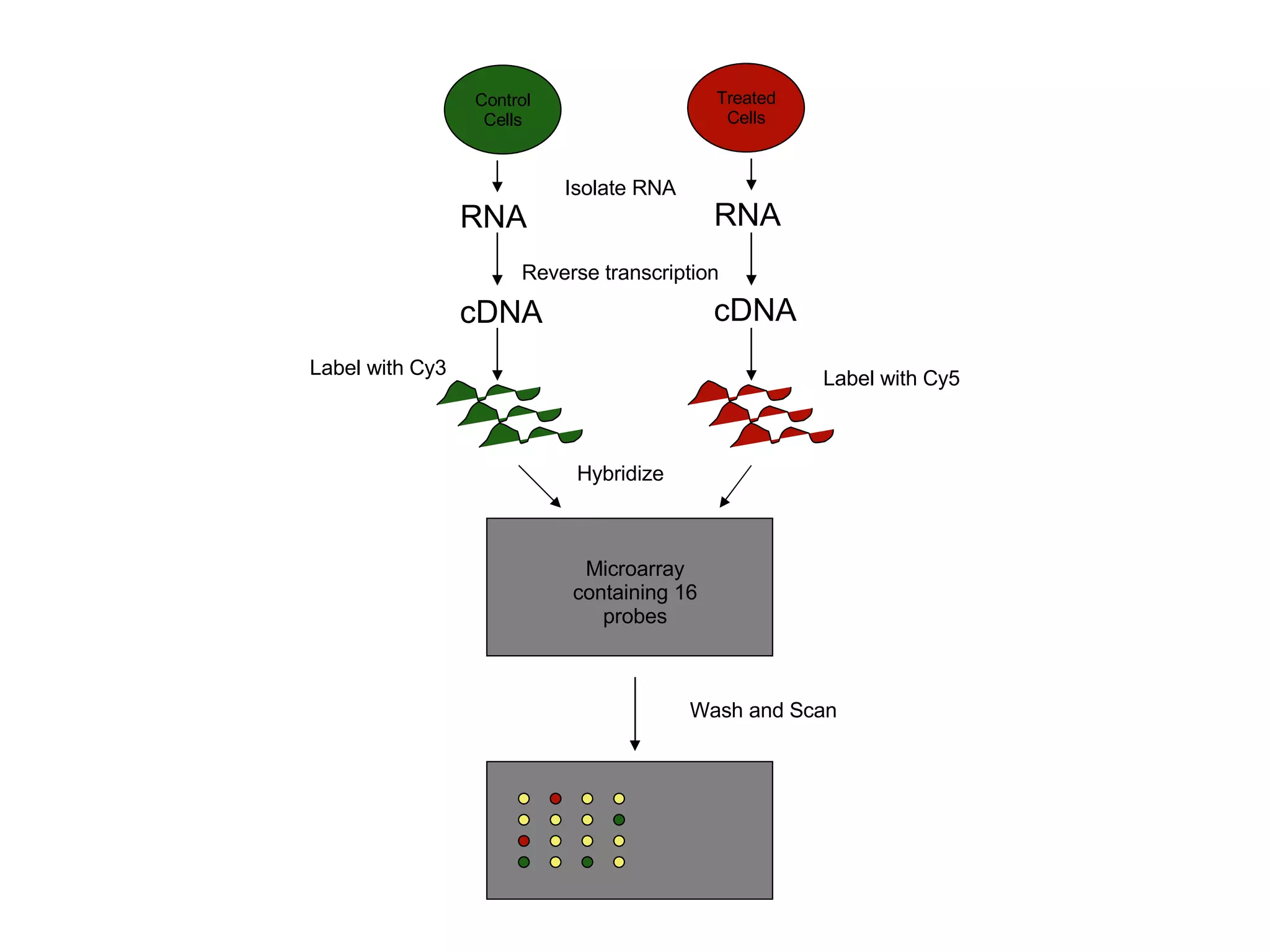
O documento descreve o que é DNA microarray e como ele funciona. DNA microarray, também conhecido como chip de DNA, permite medir o nível de transcrição de cada gene no genoma, mostrando a expressão gênica. Ele detecta mRNA hibridizado a sondas de DNA fixadas em um meio sólido como uma lâmina de vidro.