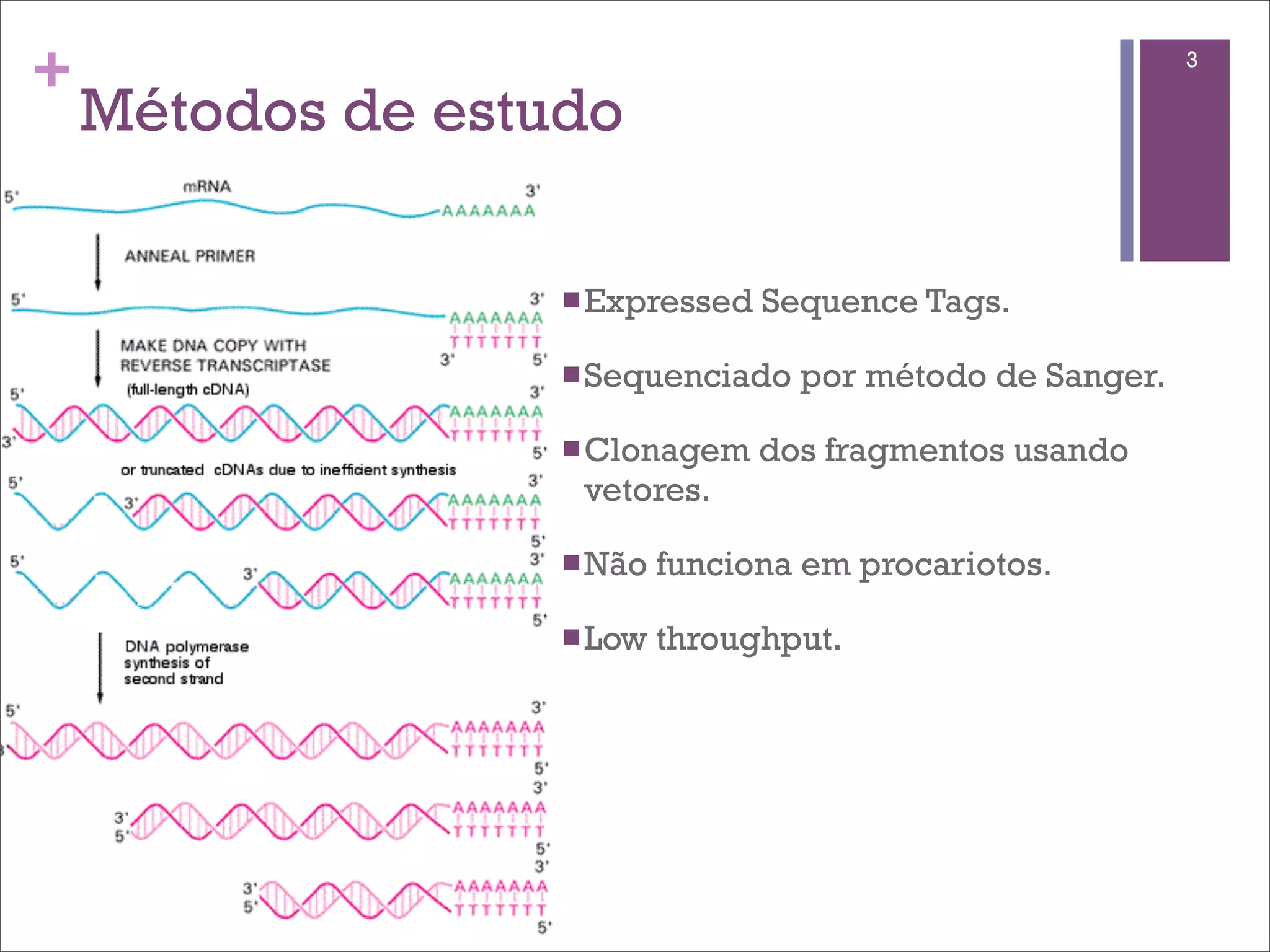
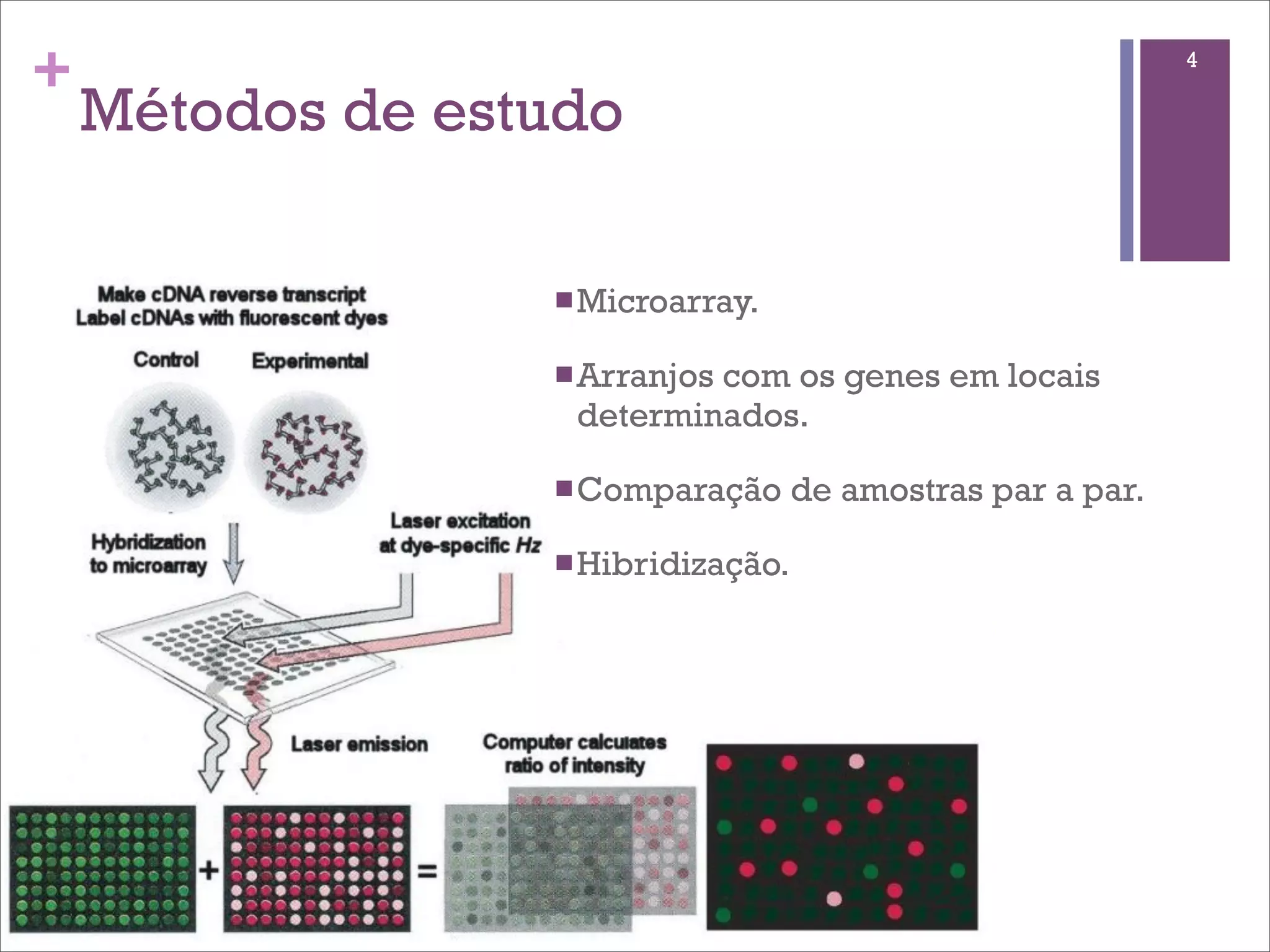
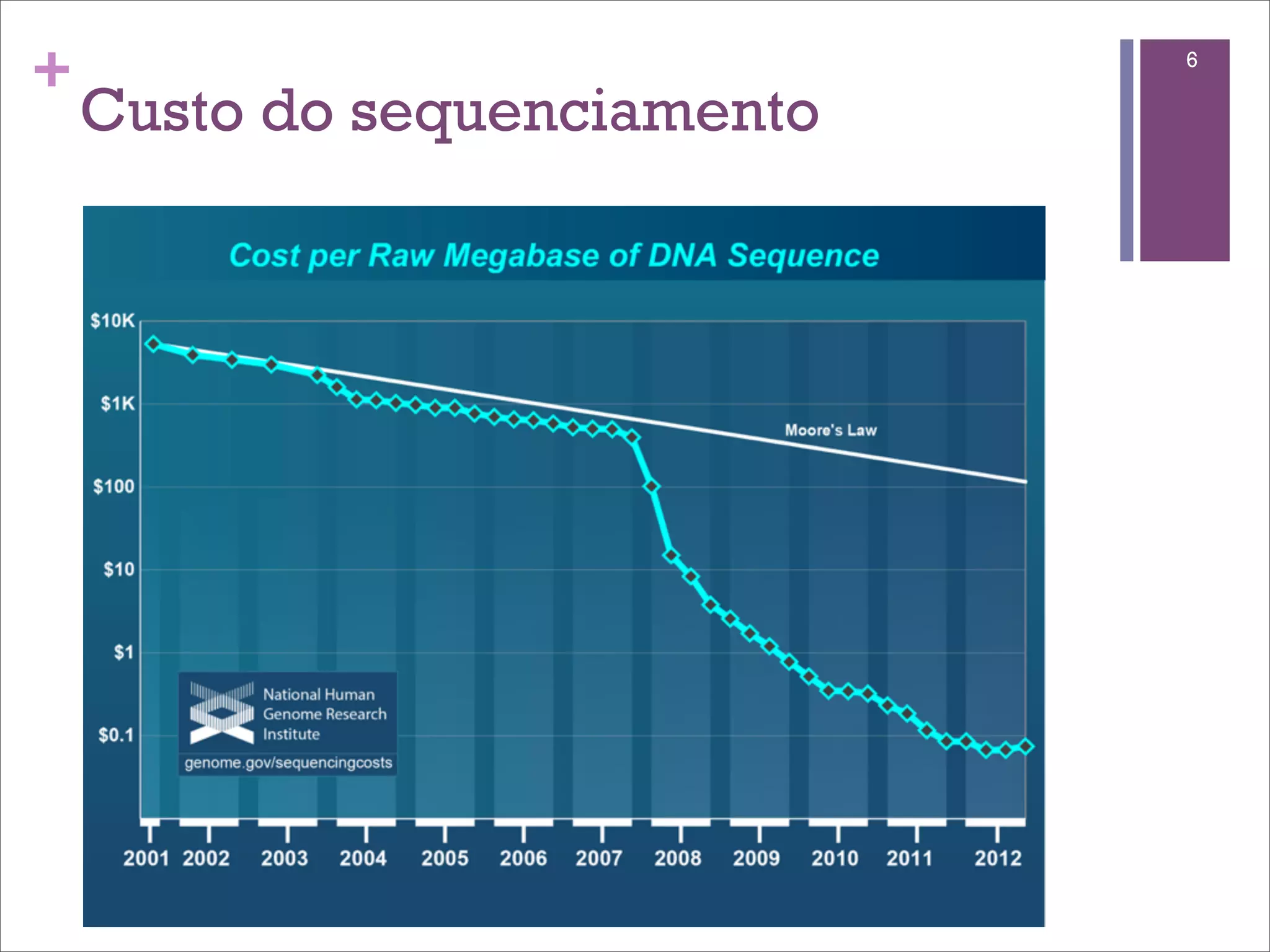
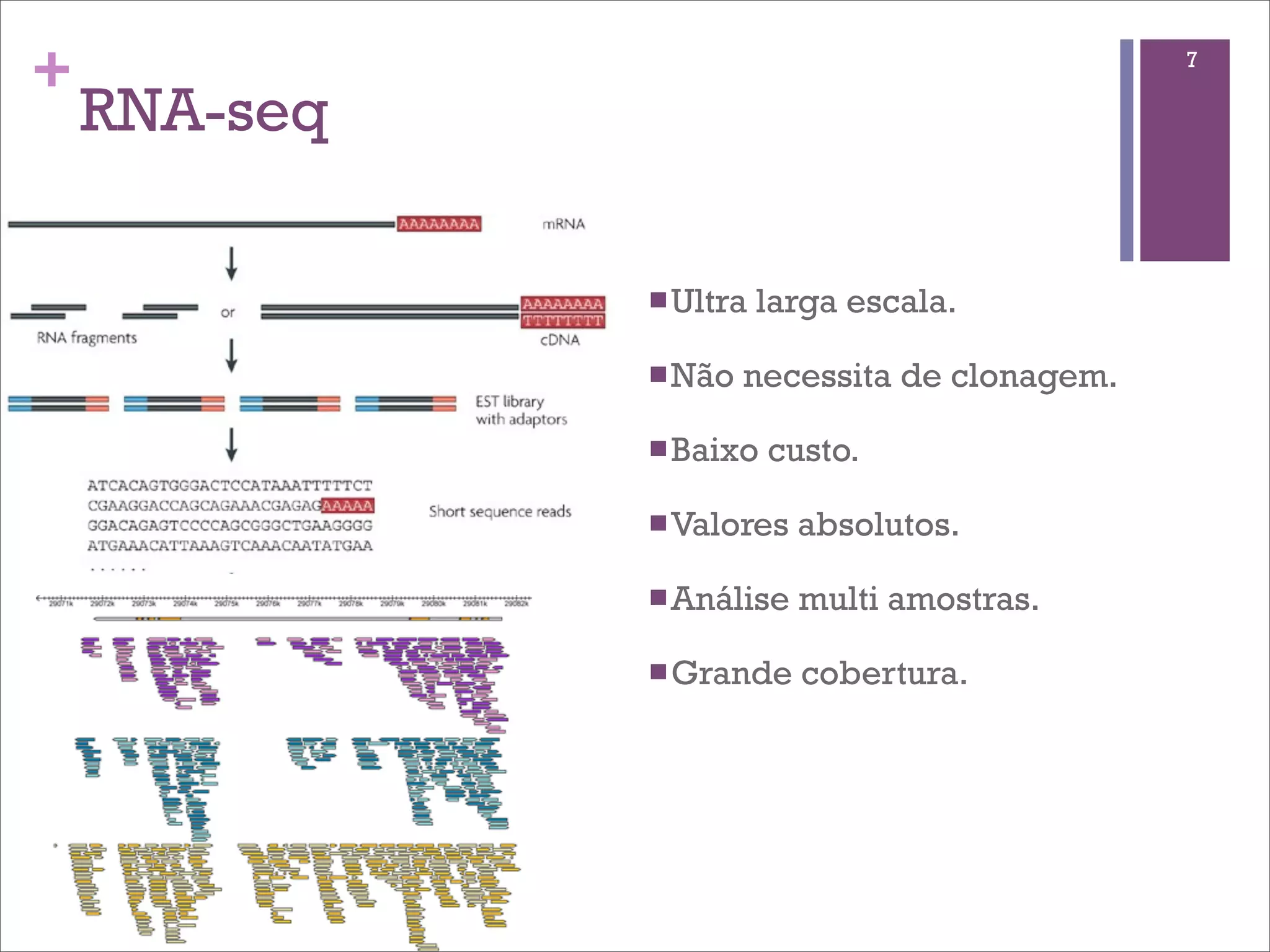
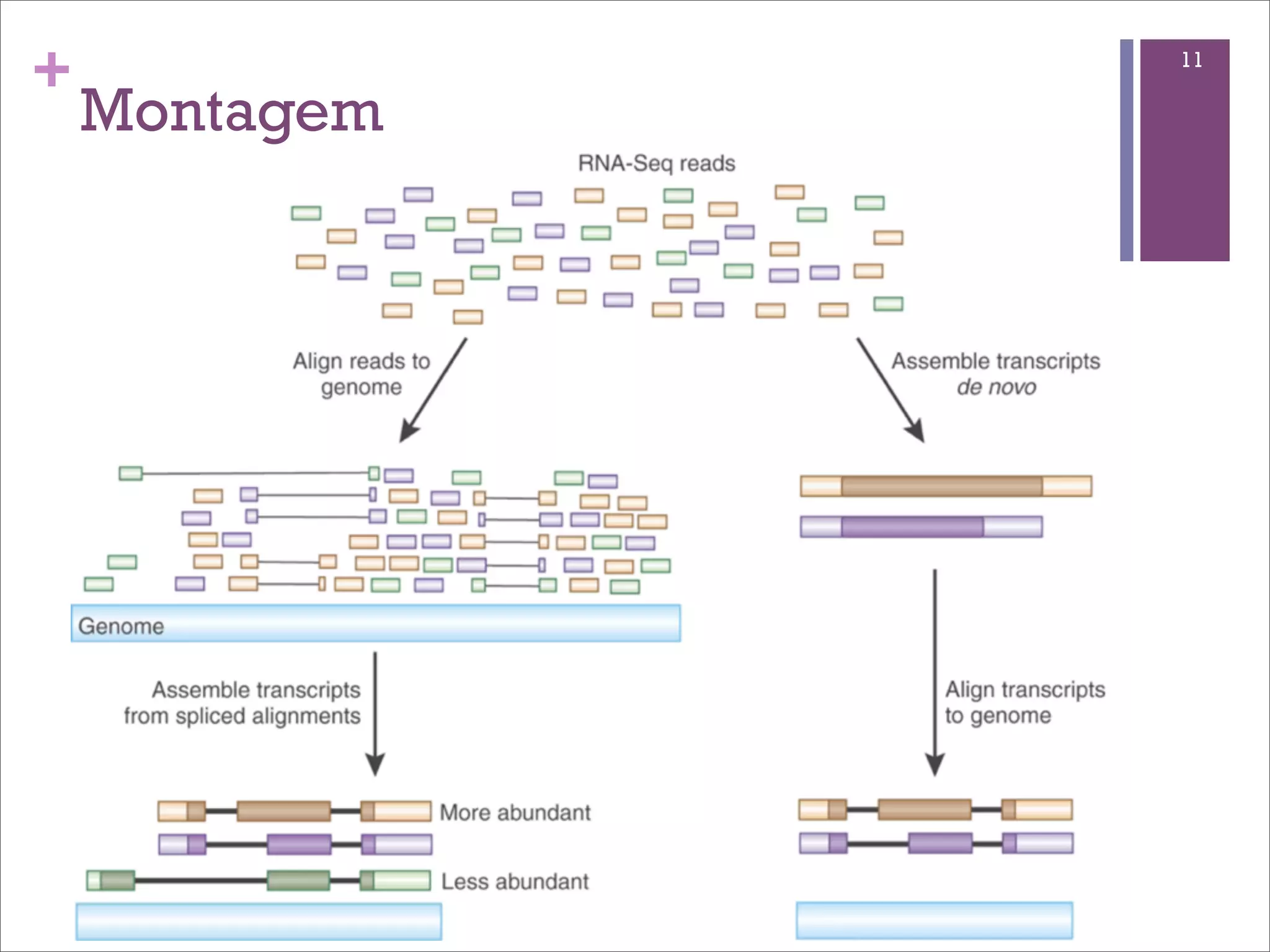
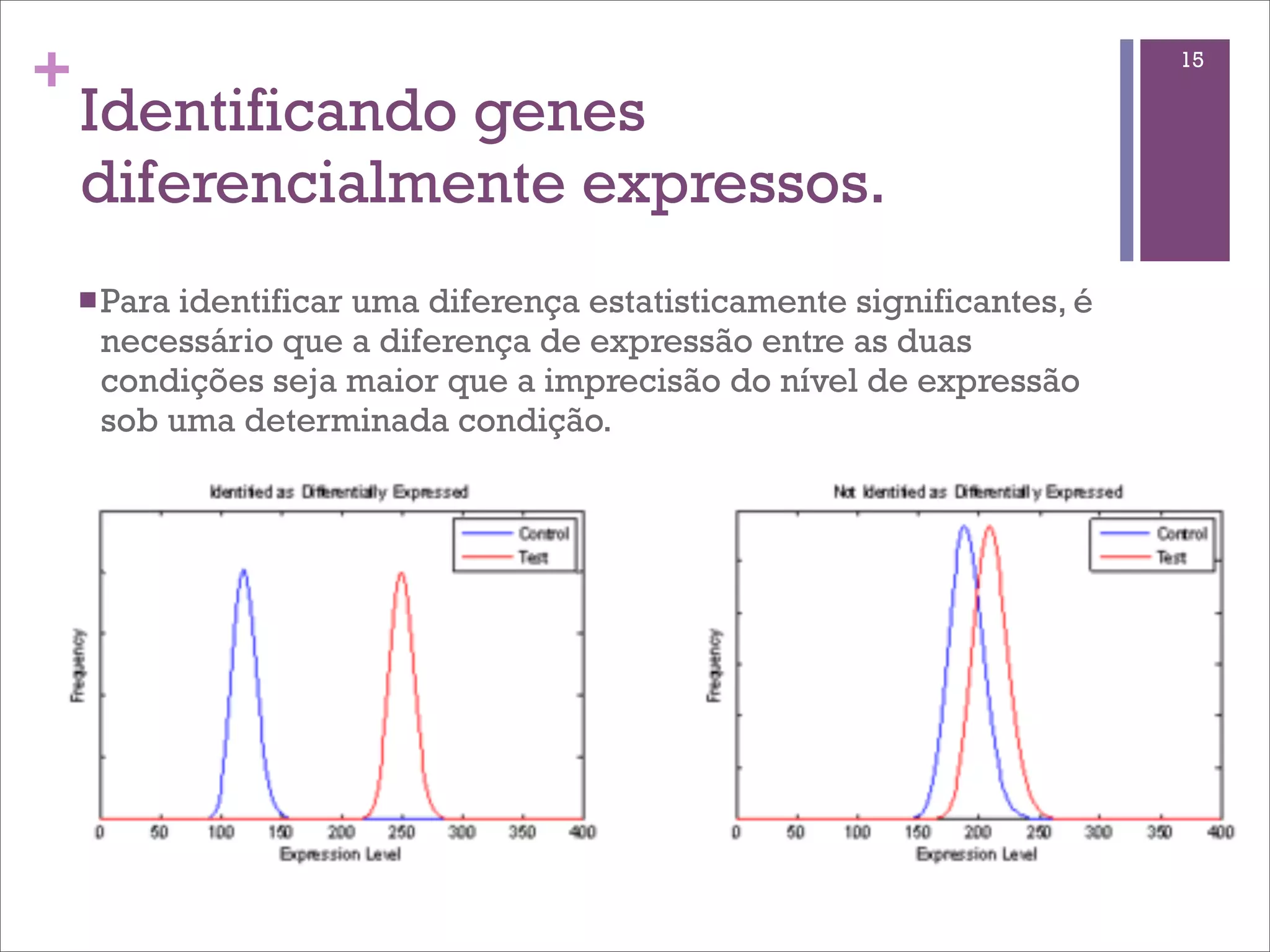
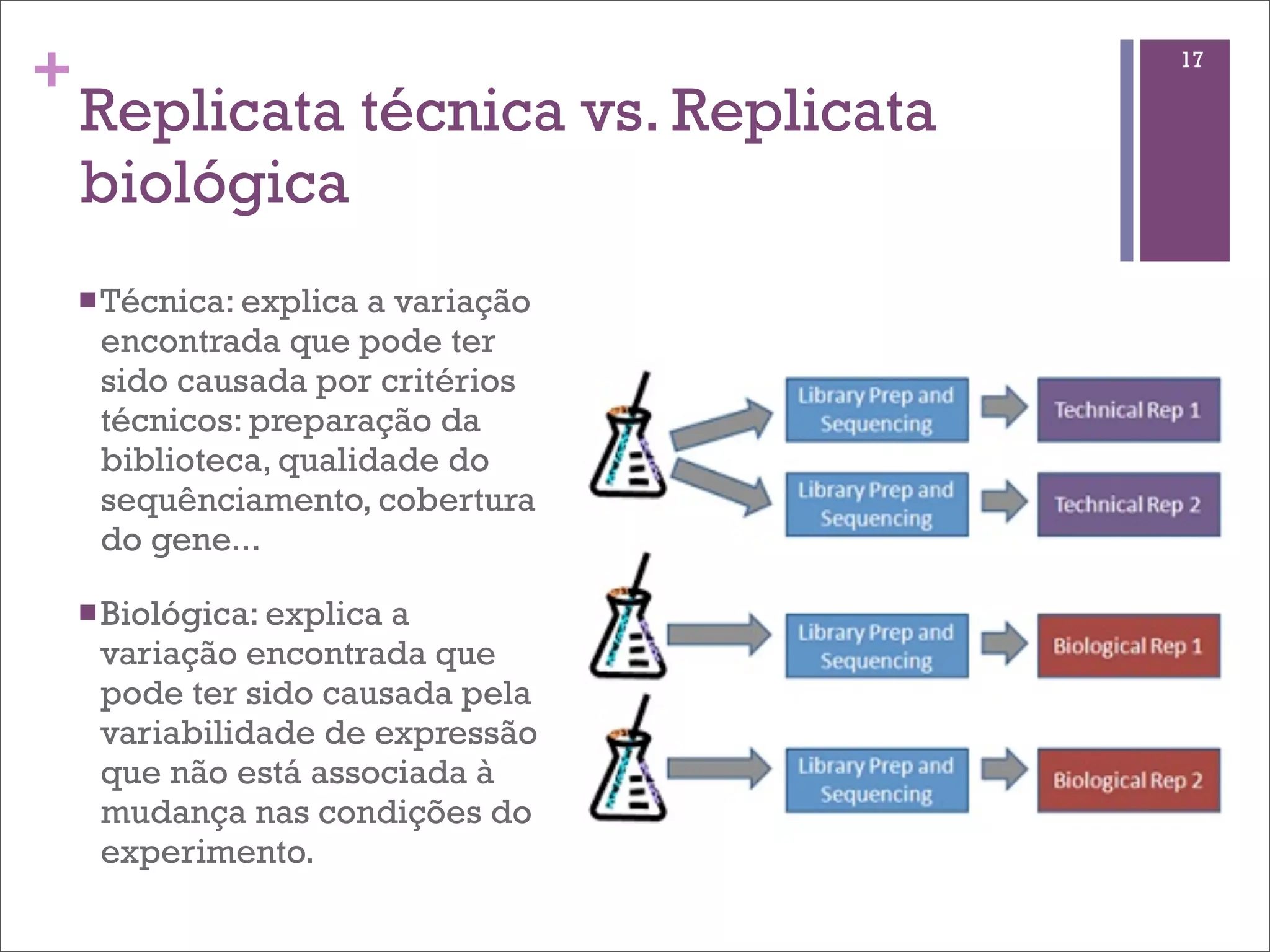
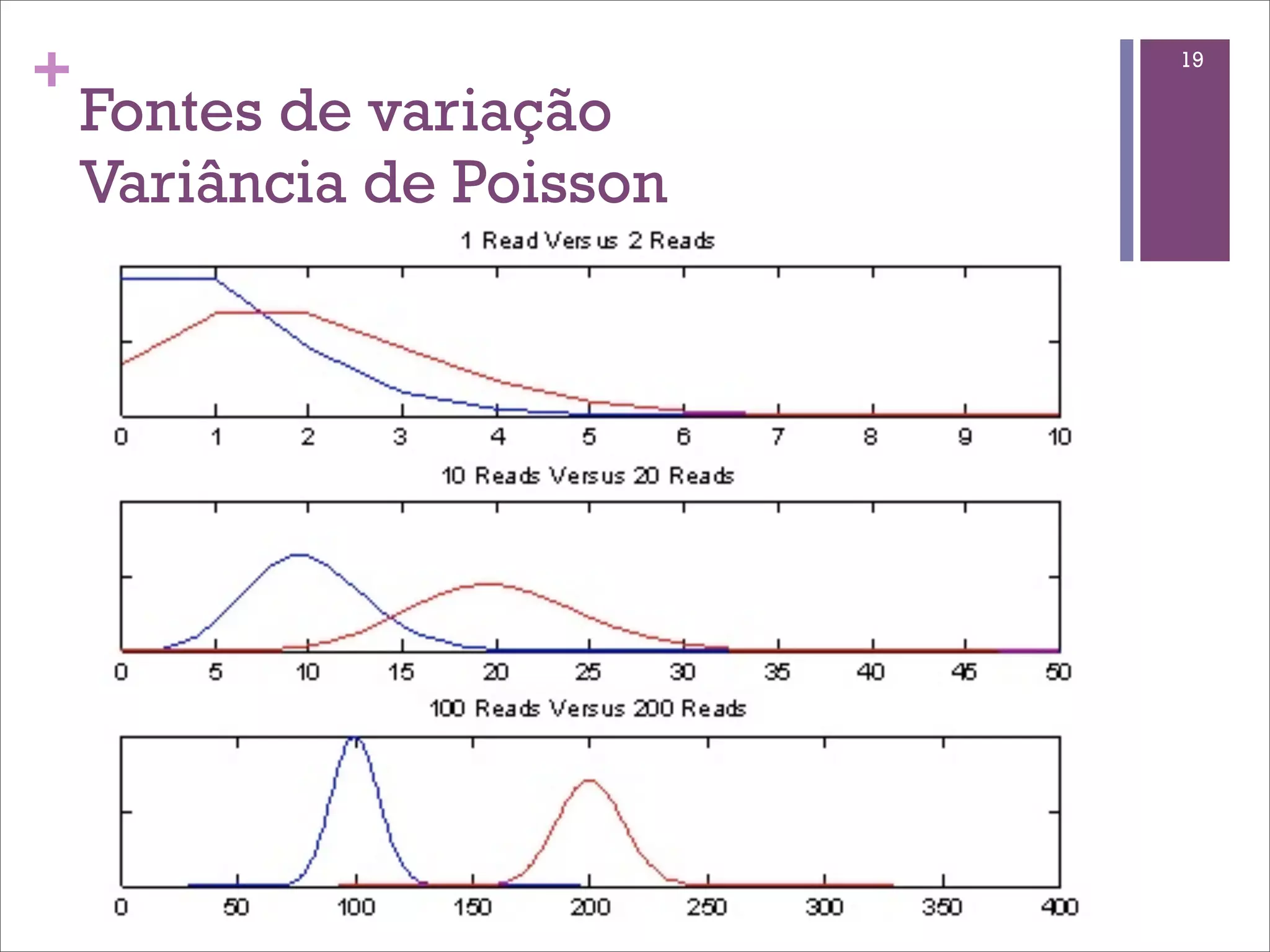
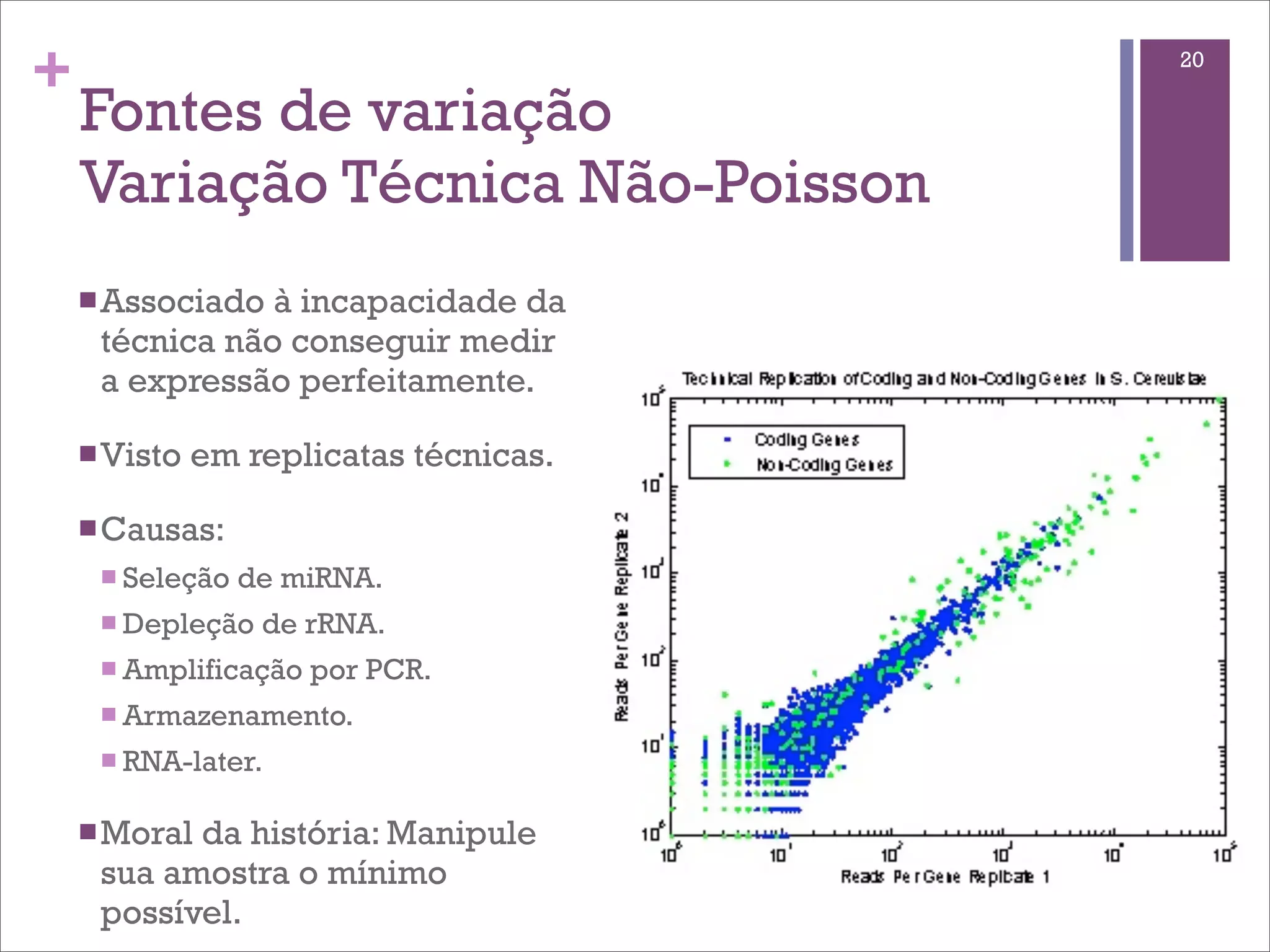
O documento discute o método de RNA-seq para estudar o transcritoma. Ele explica o que é transcritoma, métodos anteriores como ESTs e microarrays, o protocolo de RNA-seq, mapeamento de sequências, quantificação de genes, identificação de genes diferencialmente expressos e fontes de variação.