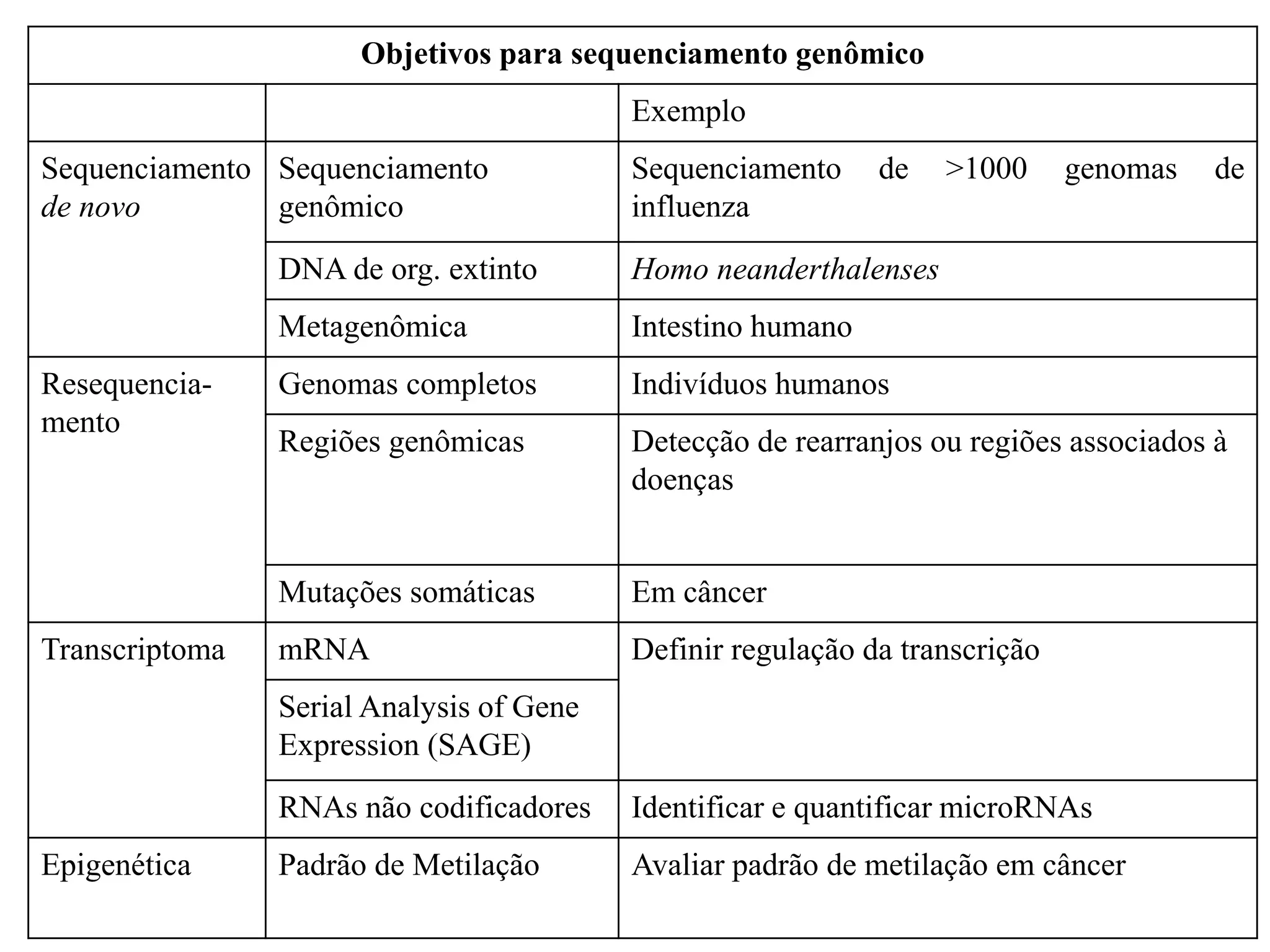
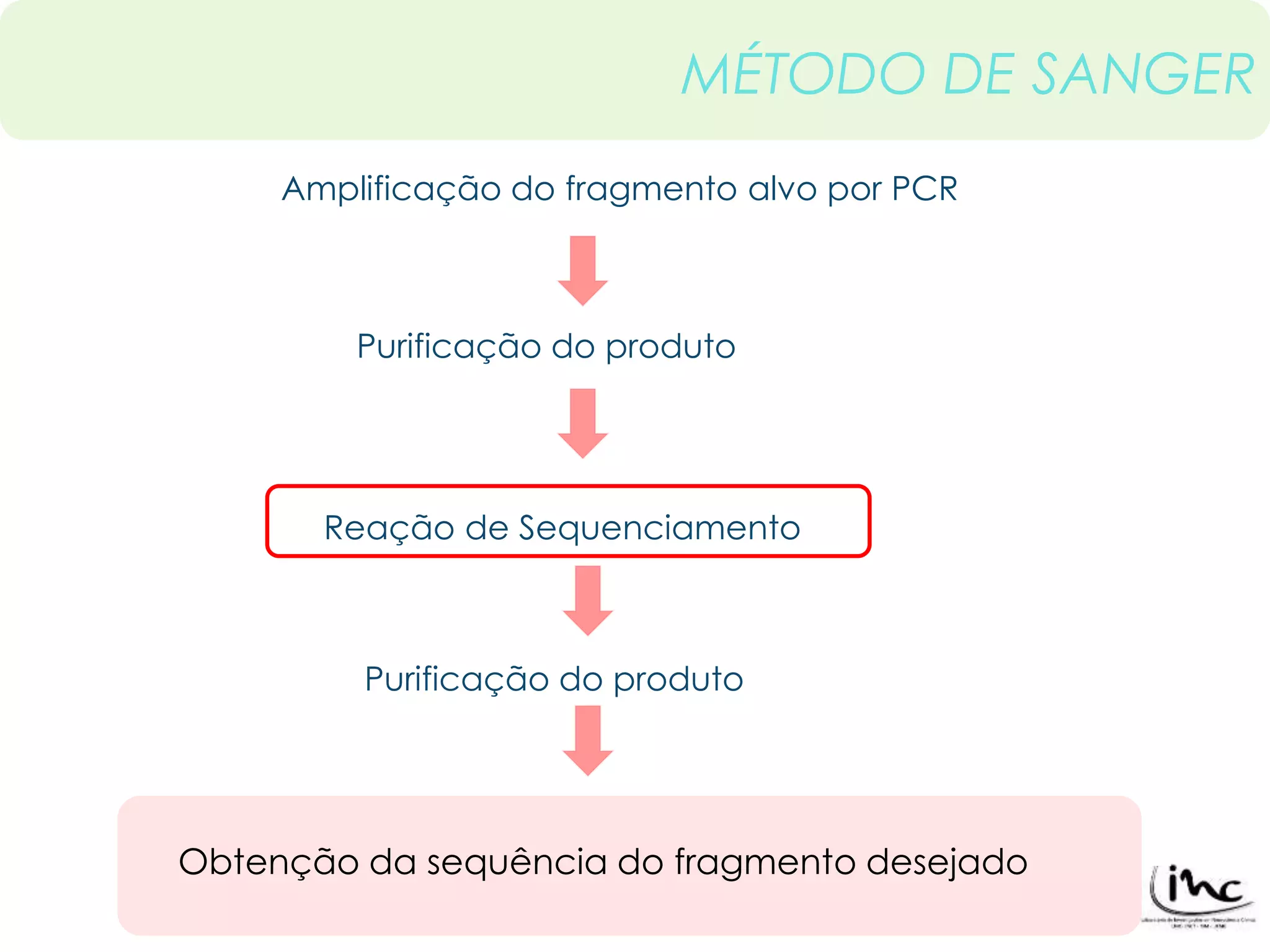
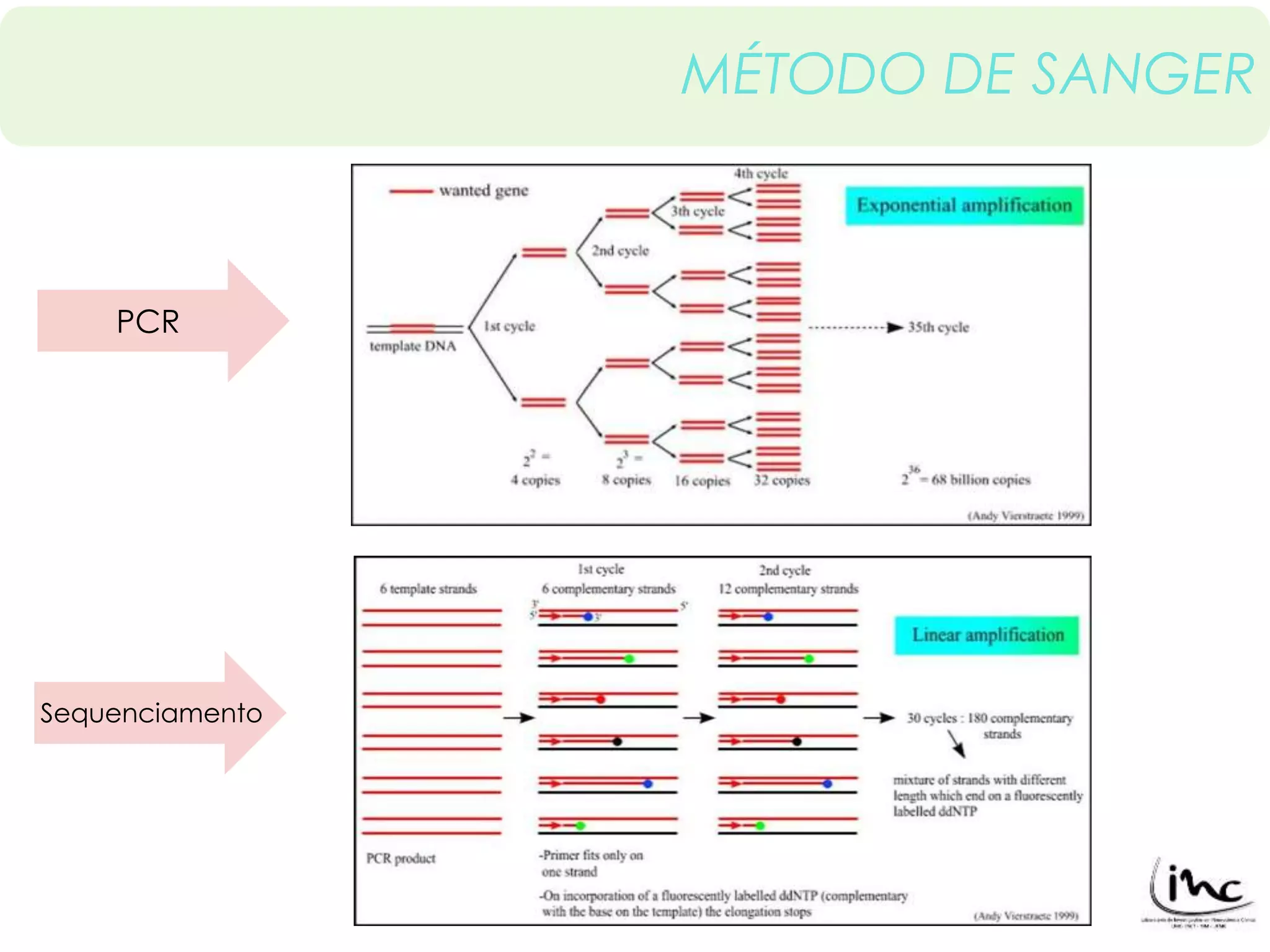
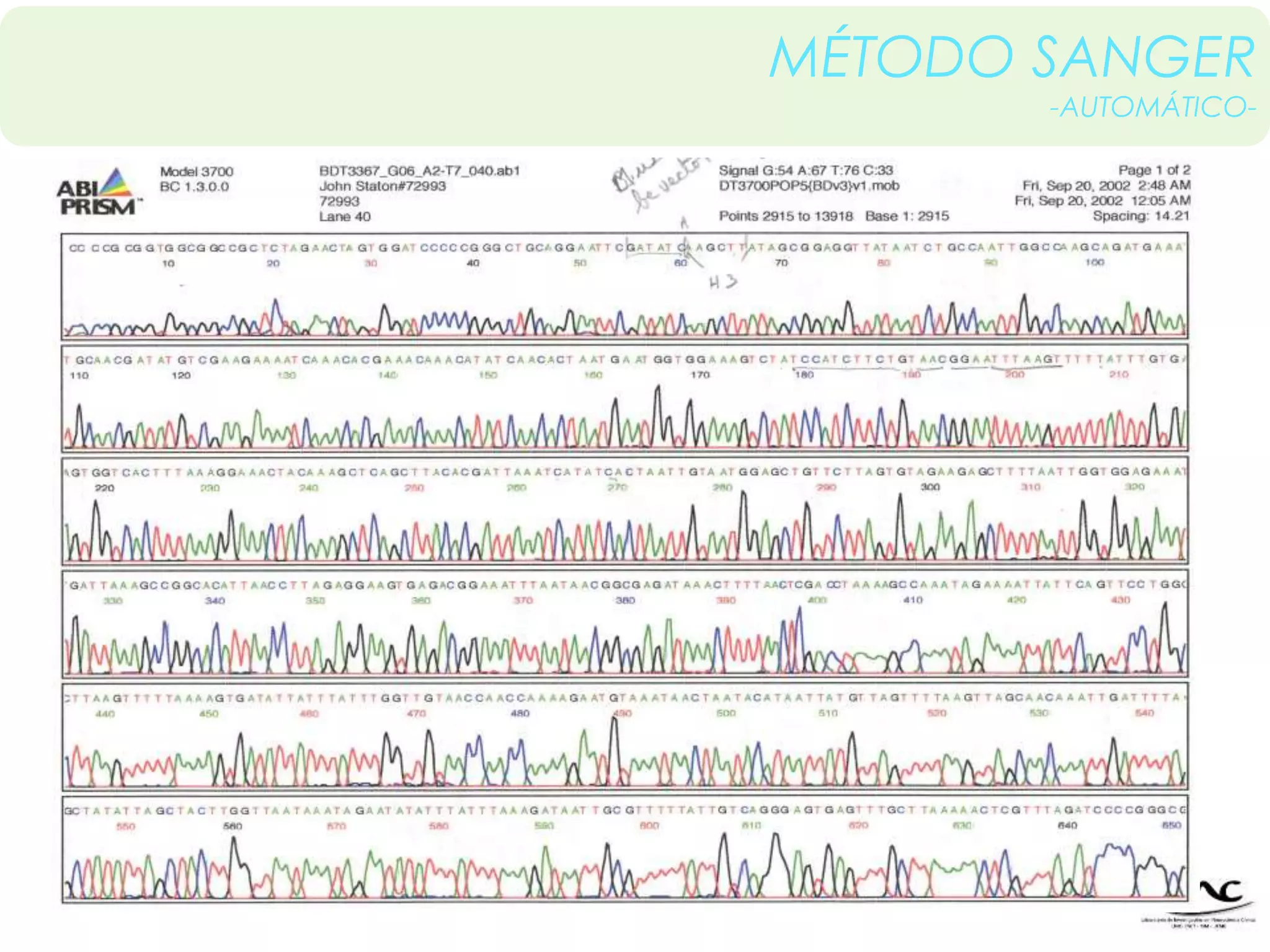
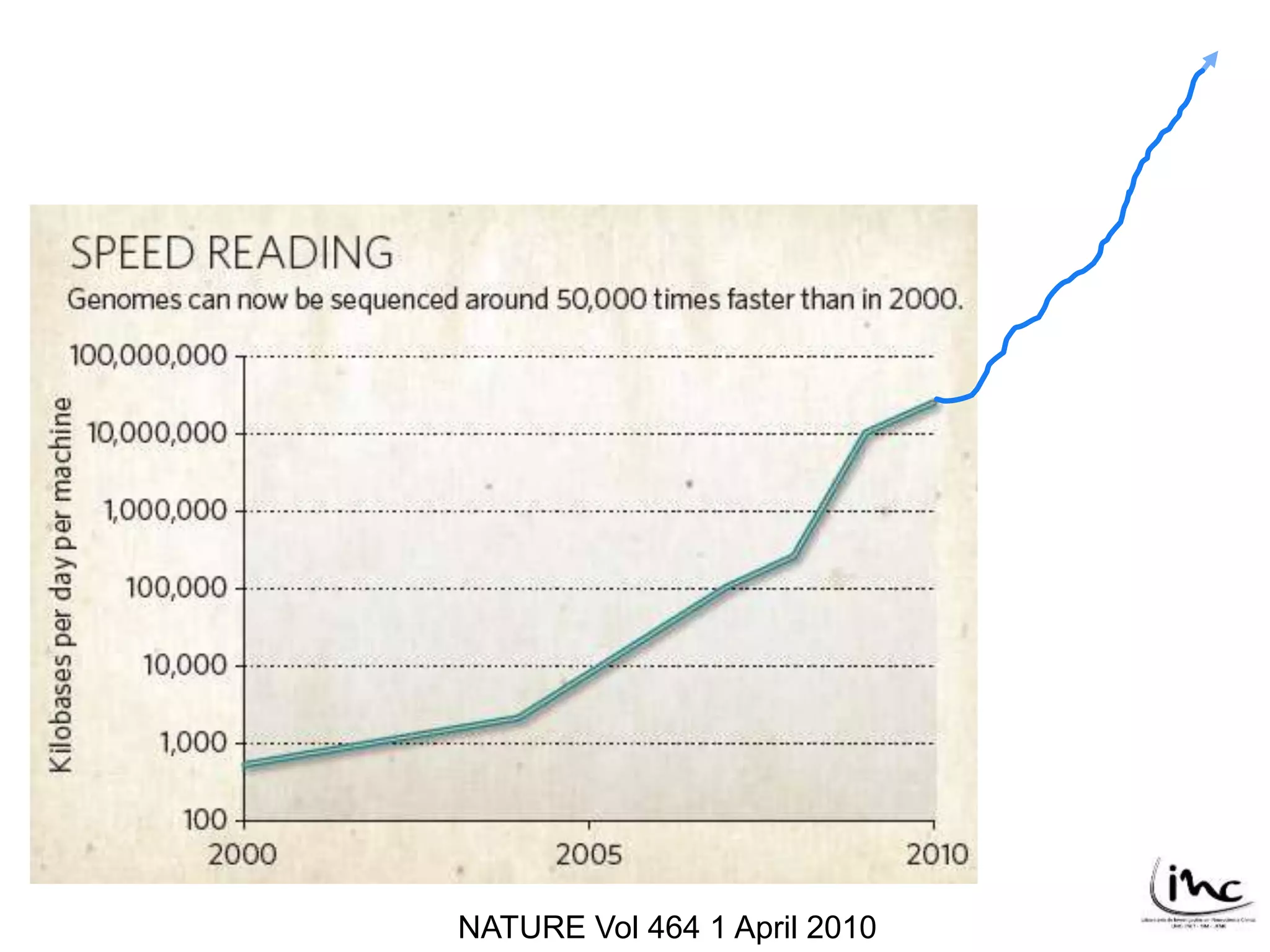
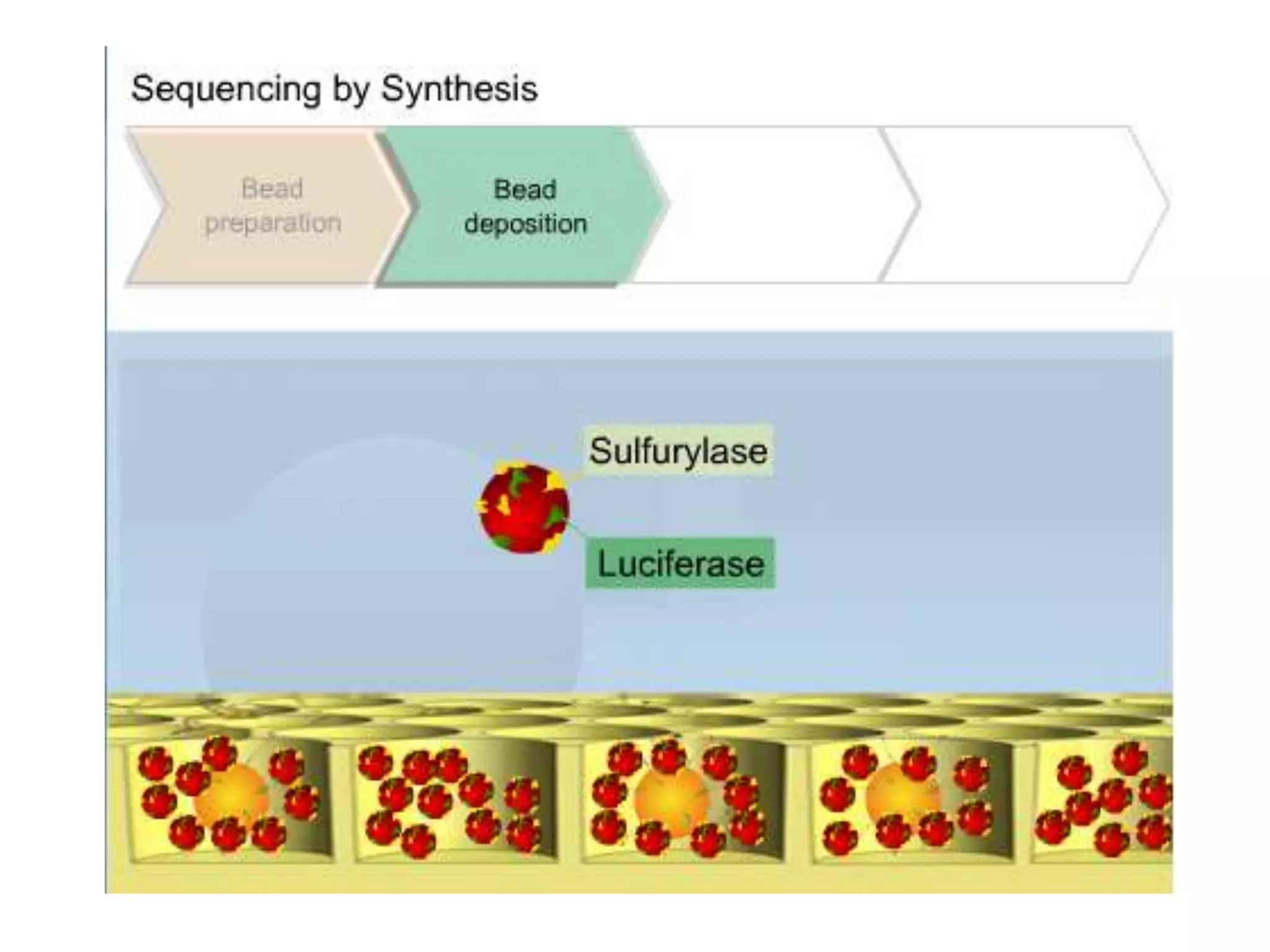
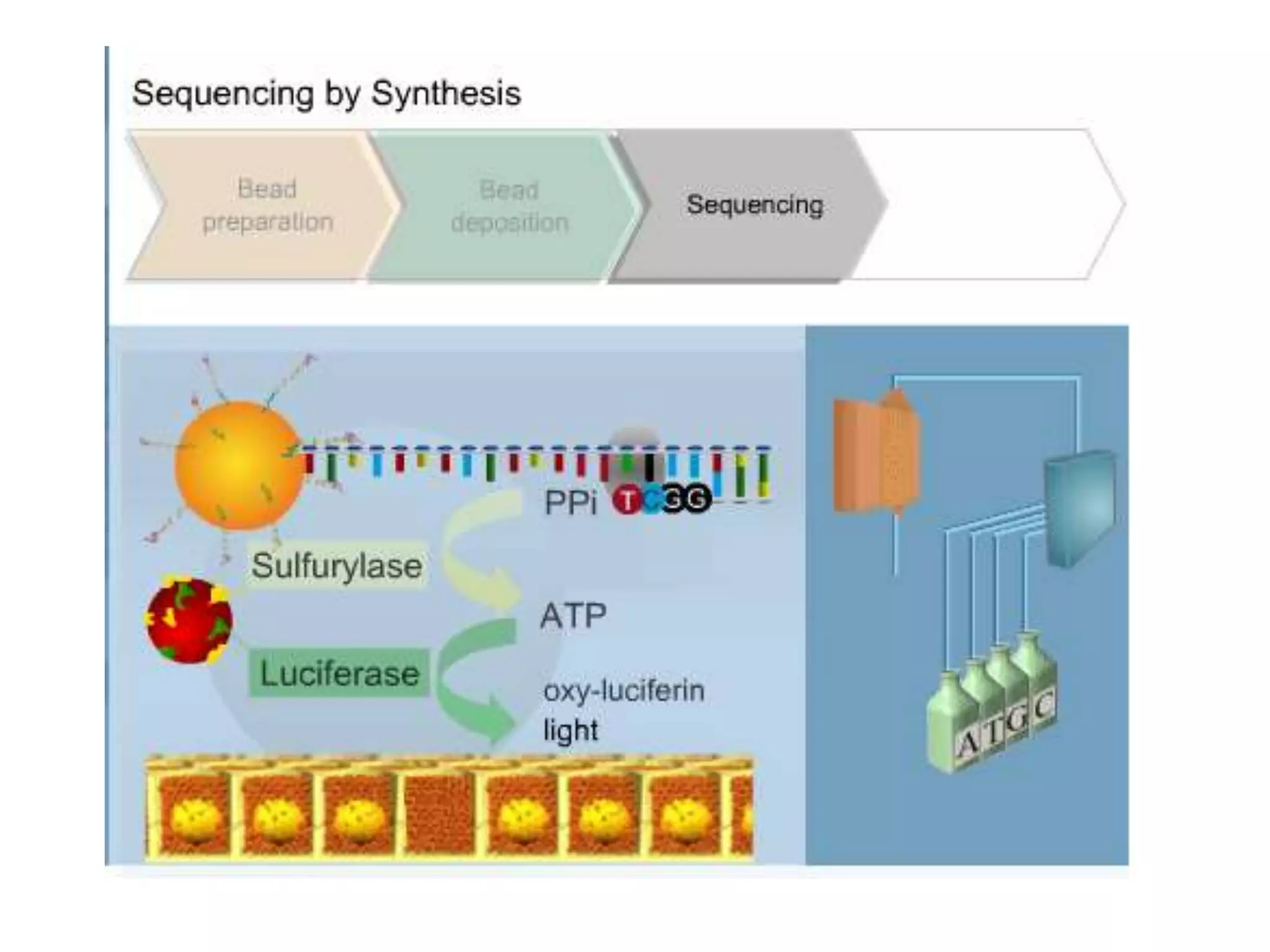
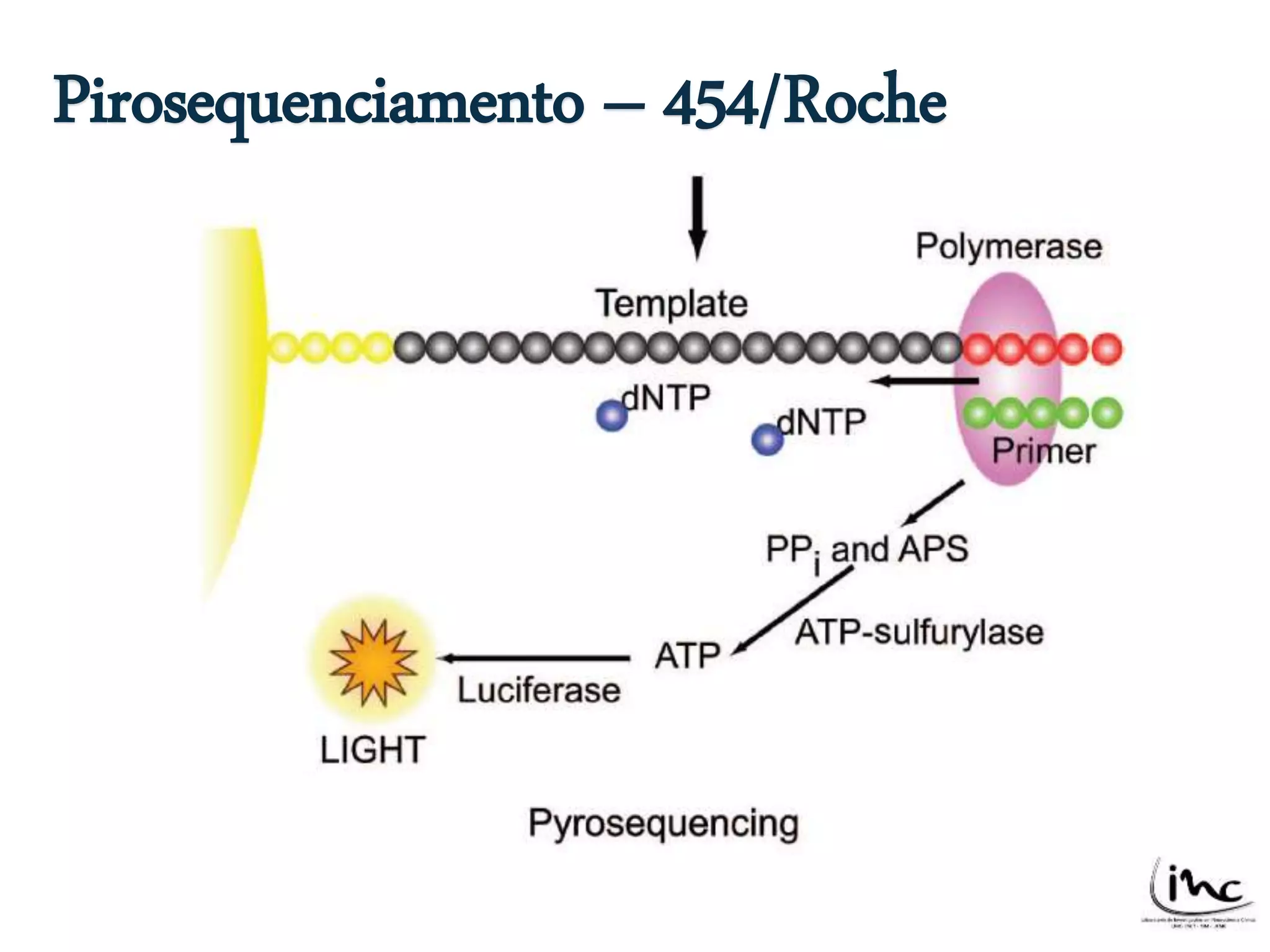
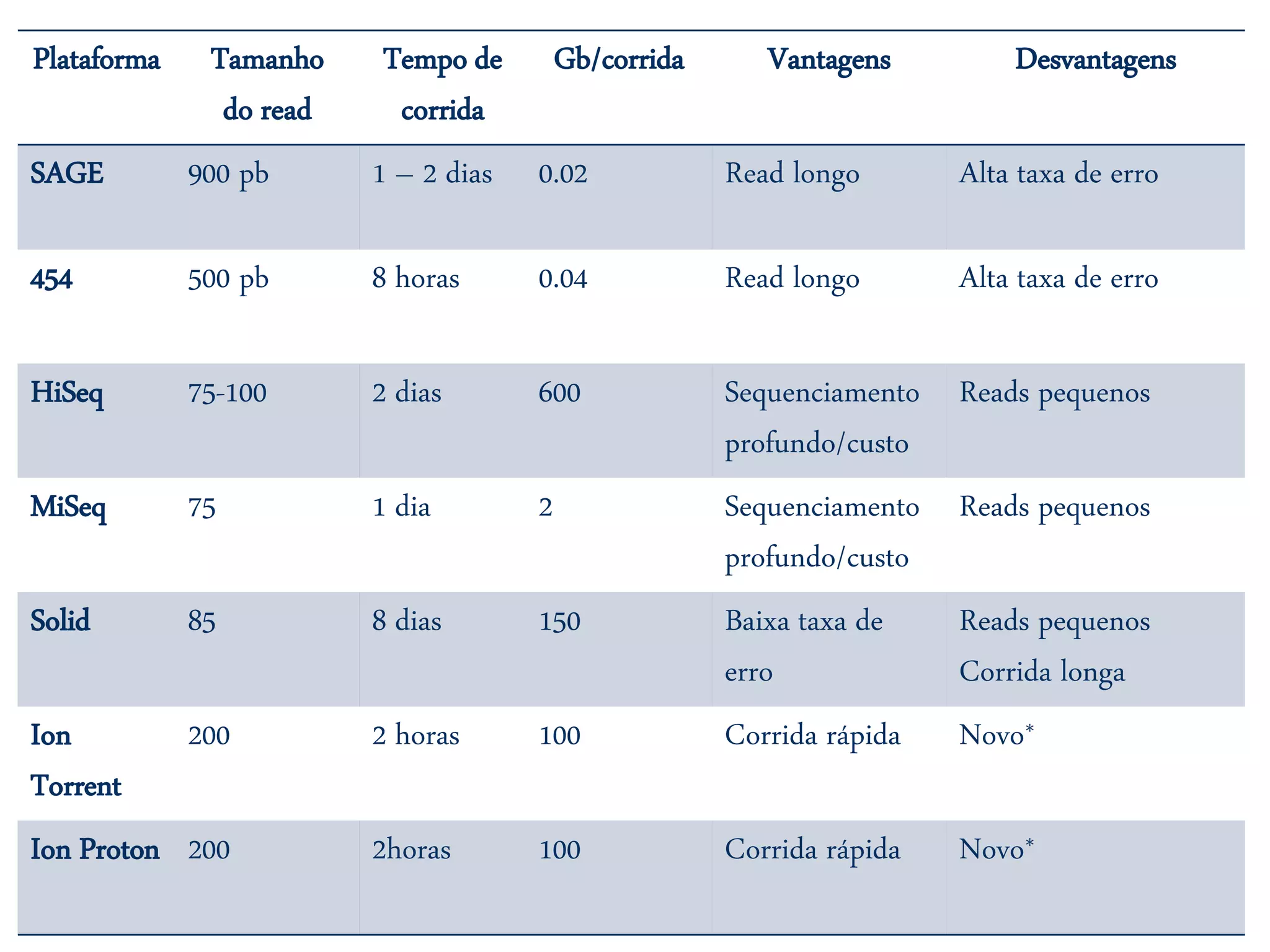
O documento discute técnicas de sequenciamento de DNA e suas aplicações. Apresenta os objetivos do minicurso, o dogma central da biologia molecular, métodos de sequenciamento como o de Sanger, e plataformas atuais como Illumina, Ion Torrent, SOLiD e suas características. Inclui também estudos de caso sobre doenças genéticas e identificação criminal por DNA.