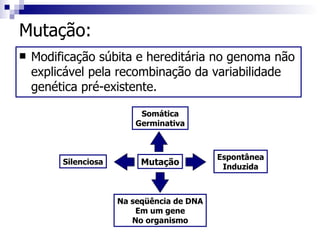
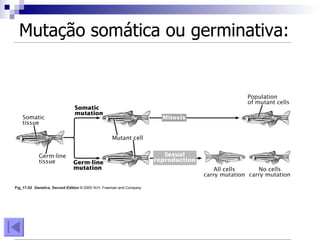
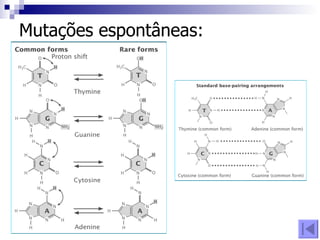
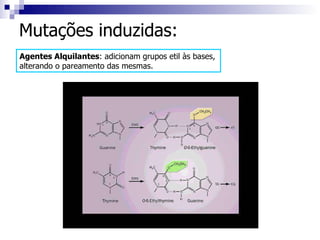
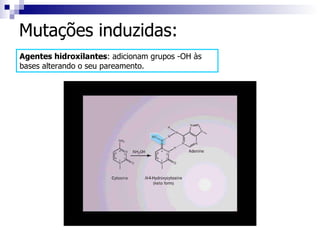
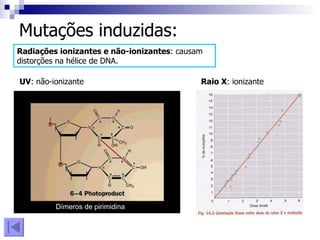
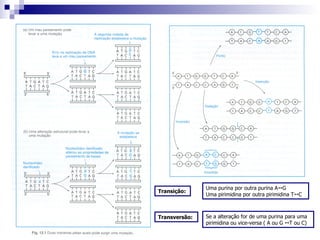
1) O documento discute mutação e reparo do material genético, incluindo causas de mutações espontâneas e induzidas e mecanismos de reparo como excisão de bases e nucleotídeos.
2) As mutações podem ocorrer na sequência do DNA ou em genes e ter efeitos neutros, alterar o fenótipo ou causar doenças.
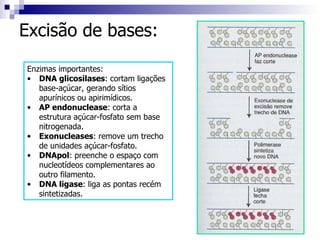
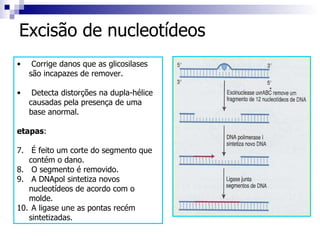
3) O reparo do DNA danificado pode ocorrer por reversão direta, excisão de bases ou nucleotídeos para remover danos e sintetizar