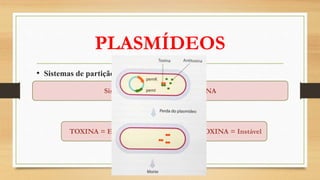
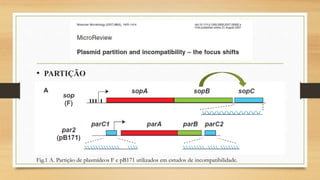
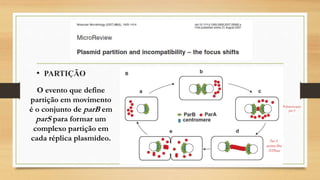
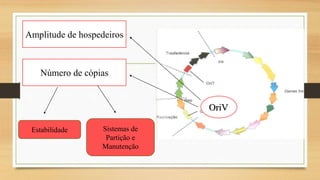
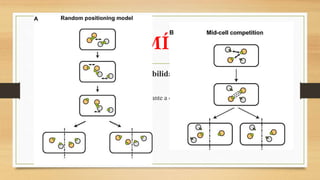
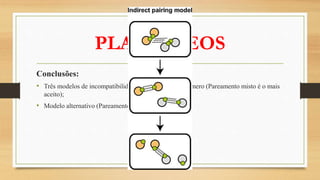
O documento descreve as principais características dos plasmídeos, incluindo sua definição, tamanho, mecanismos de replicação, controle do número de cópias, classificação de acordo com a propriedade de transferência, sistemas de partição e manutenção e grupos de incompatibilidade.