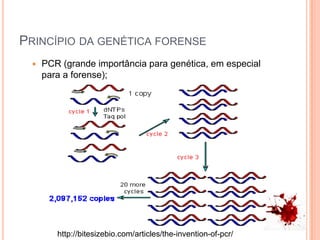
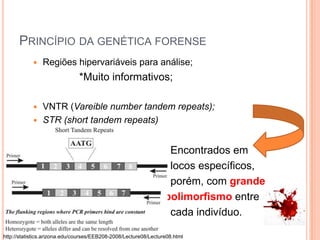
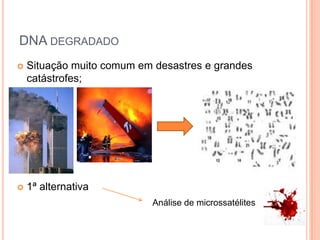
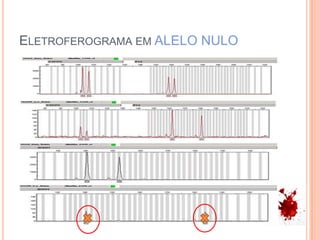
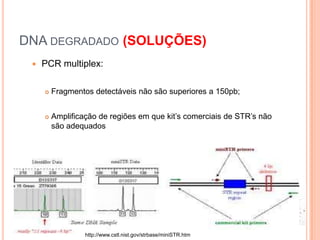
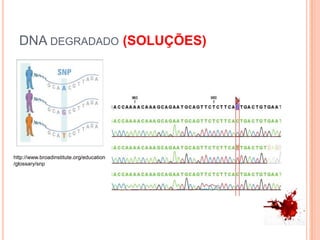
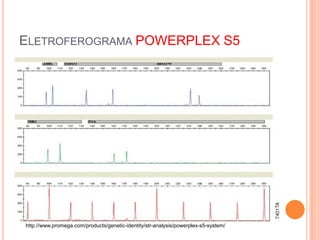
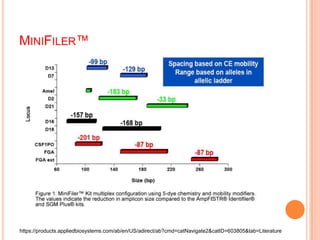
O documento aborda técnicas de análise de DNA degradado em genética forense, destacando a importância da amplificação por PCR e o uso de diferentes marcadores, como STRs e SNPs, para identificação humana. As limitações do DNA degradado, frequentemente encontrado em cenas de crime, são apresentadas junto com soluções, como o uso de mini-STRs e DNA mitocondrial. O texto também menciona kits disponíveis para a análise eficaz de amostras comprometidas.