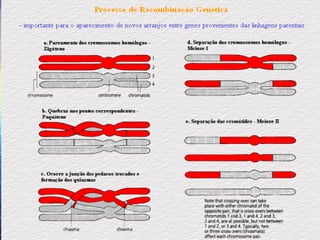
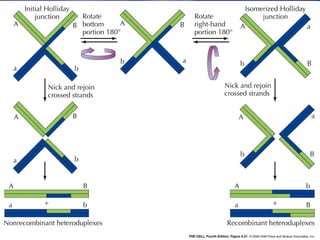
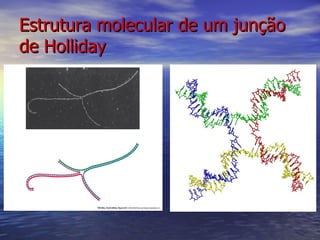
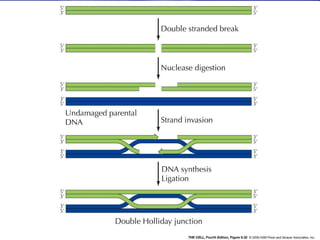
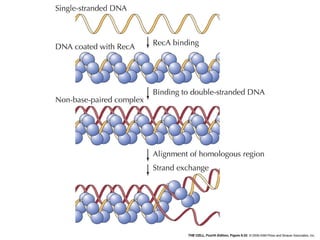
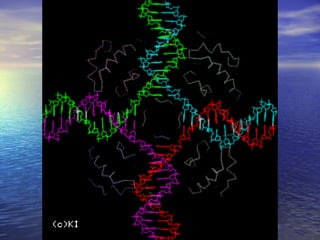
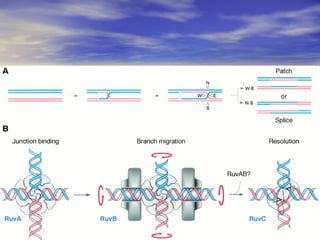
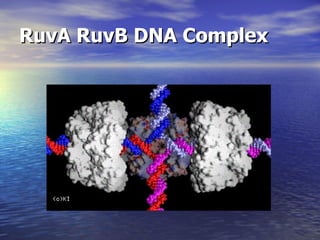
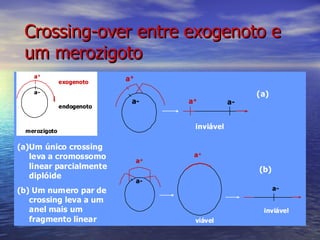
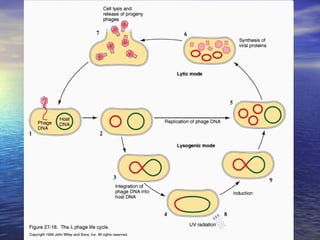
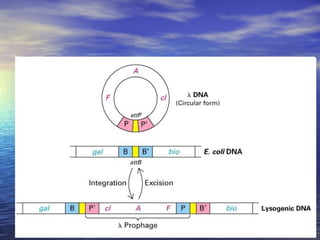
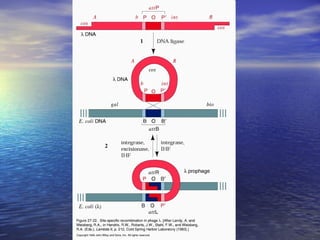
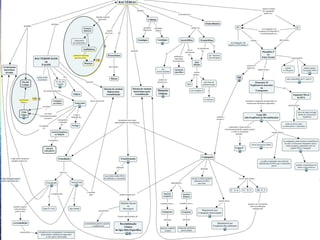
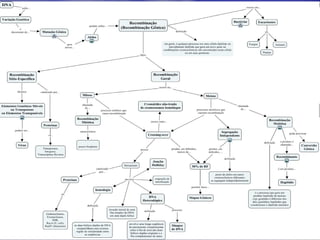
(1) A recombinação genética envolve a troca de material genético entre cromossomos homólogos durante a meiose, gerando diversidade genética. (2) Na meiose, ocorre o crossing-over entre cromossomos homólogos, resultando na formação de cromossomos recombinantes. (3) Vários mecanismos moleculares, como o modelo de Holliday, explicam como ocorre a recombinação genética ao nível das moléculas de DNA.