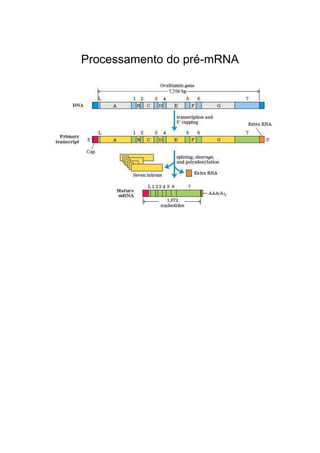
O pré-mRNA eucariótico passa por diversas modificações após a transcrição, incluindo a remoção de íntrons, adição de um cap 5' e cauda poli(A), e pode sofrer edição. Isto gera o mRNA maduro, que é transportado para o citoplasma e traduzido em proteínas. Já o mRNA procariótico sofre poucas modificações e a transcrição e tradução ocorrem simultaneamente.