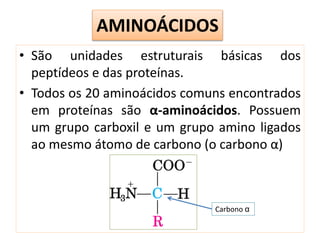
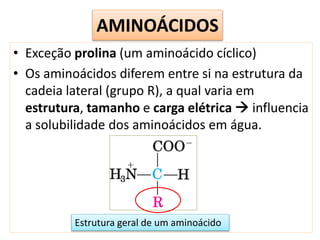
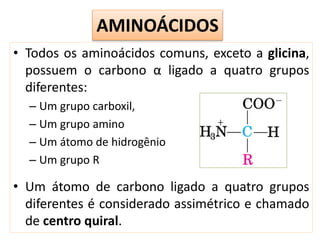
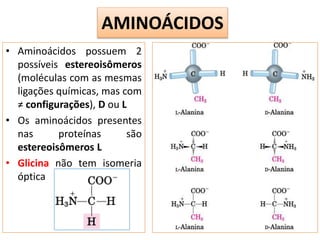
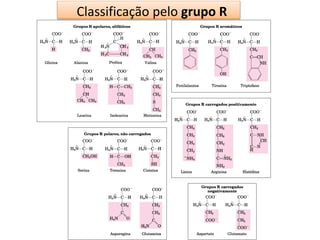
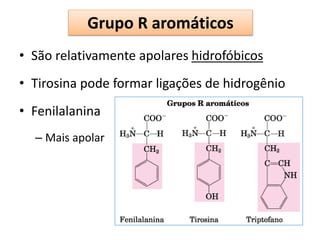
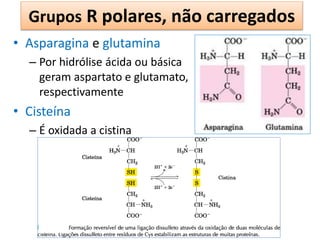
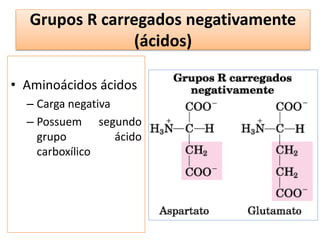
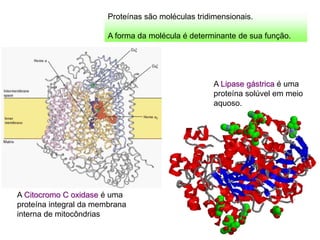
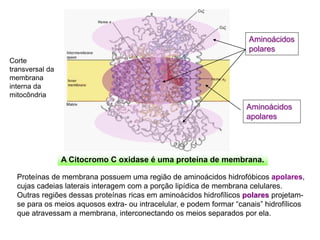
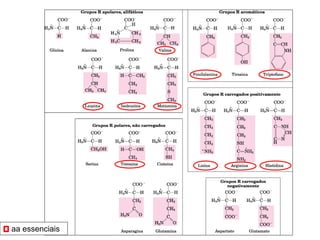
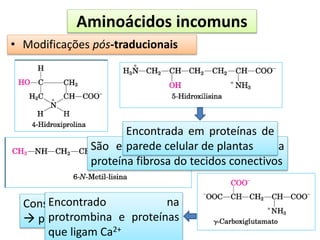
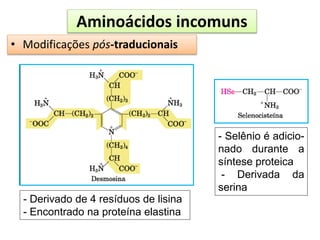
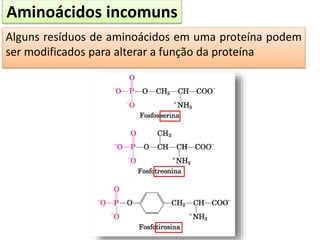
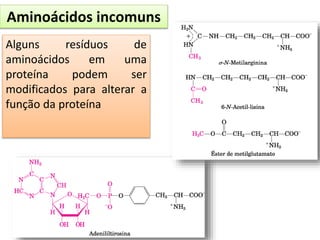
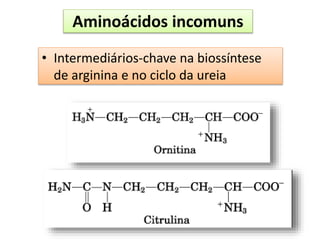
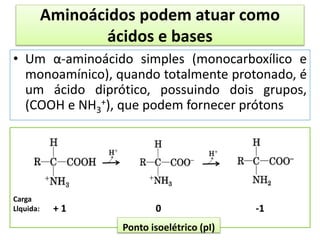
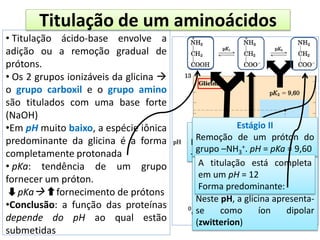
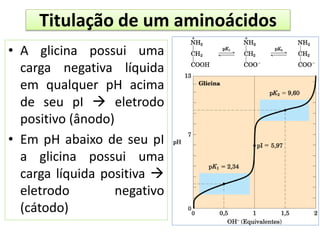
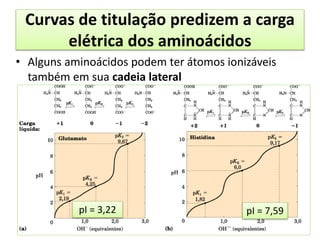
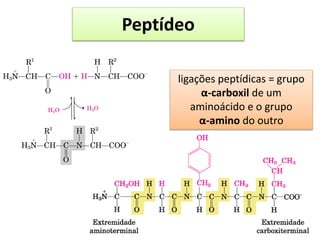
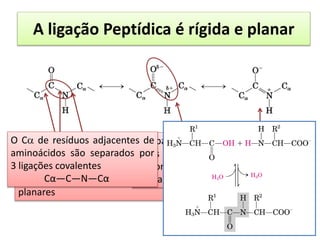
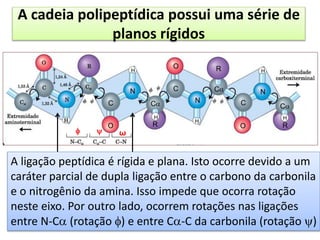
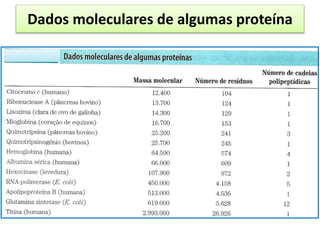
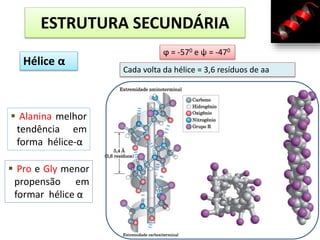
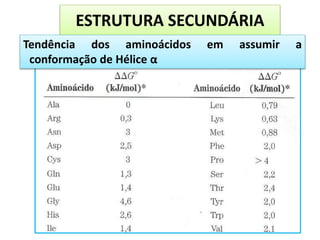
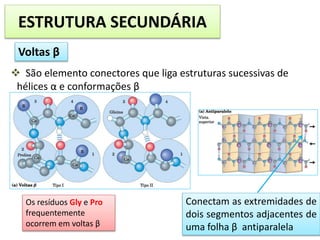
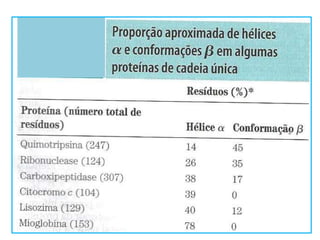
O documento discute os aminoácidos, peptídeos e proteínas, abordando:
1) As características químicas dos aminoácidos e suas classificações;
2) A formação de ligações peptídicas na união de aminoácidos em cadeias polipeptídicas;
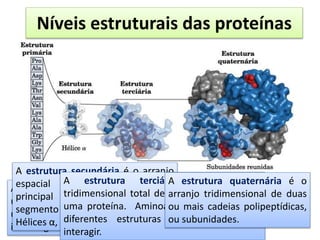
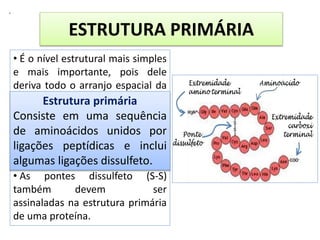
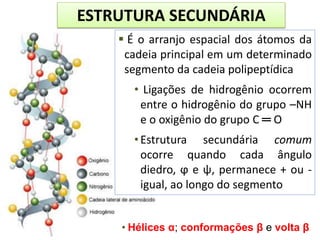
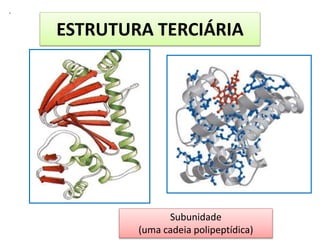
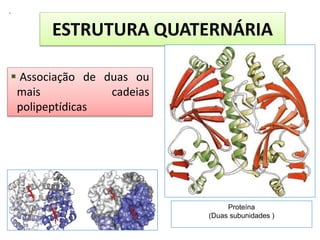
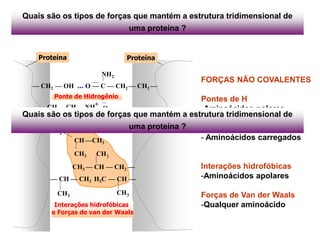
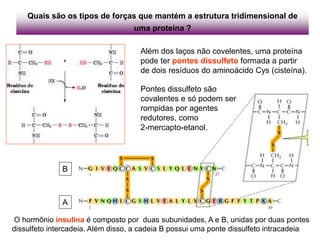
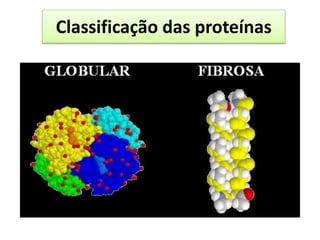
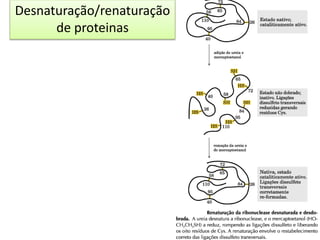
3) Os diferentes níveis estruturais das proteínas, incluindo estrutura primária, secundária, terciária e quaternária.