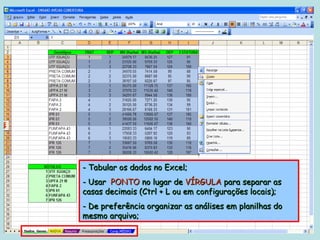
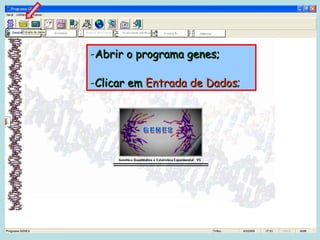
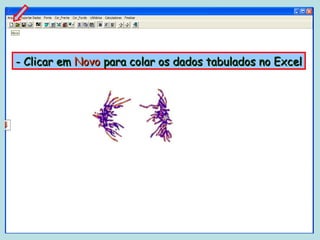
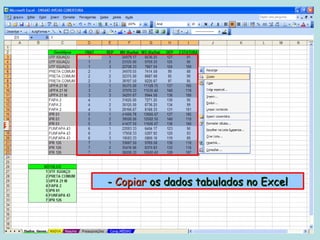
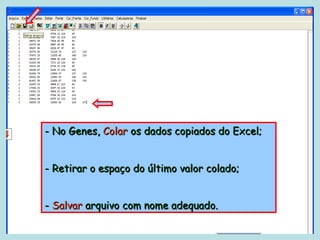
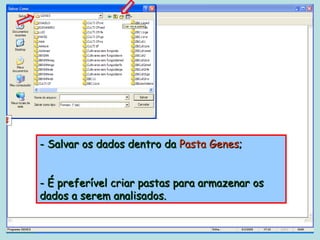
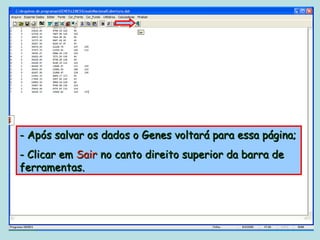
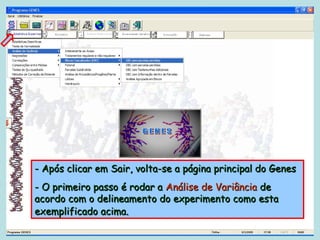
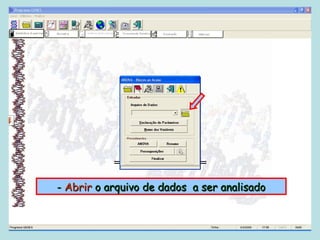
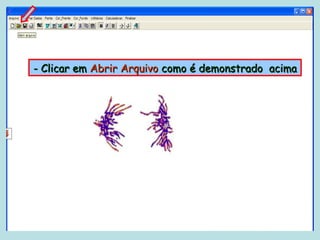
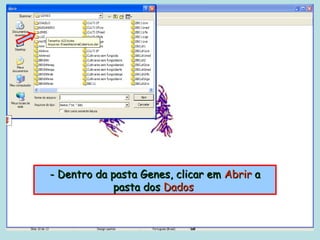
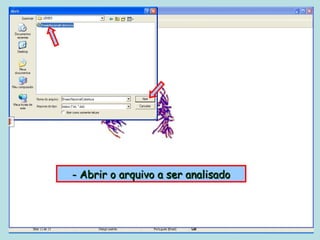
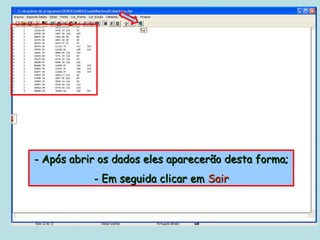
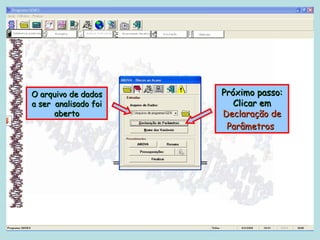
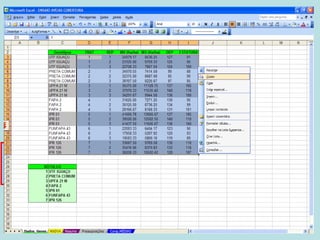
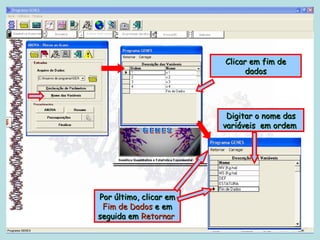
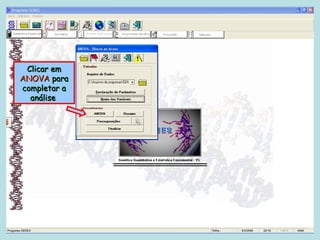
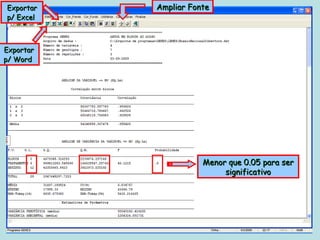
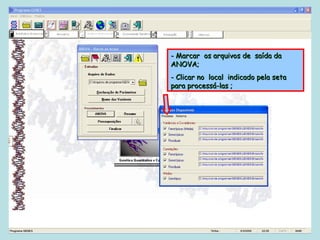
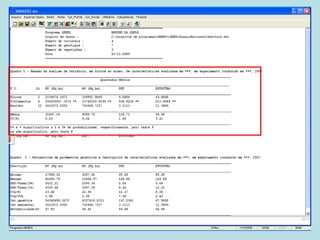
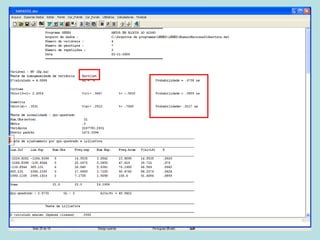
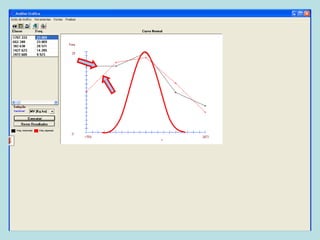
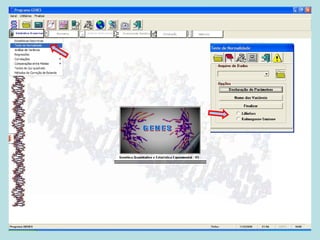
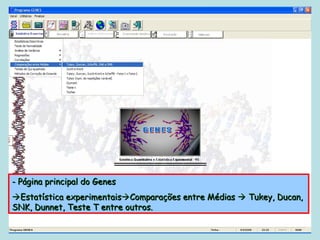
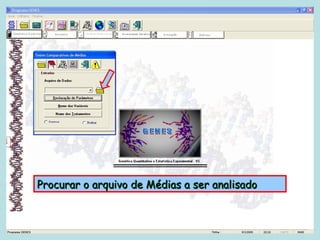
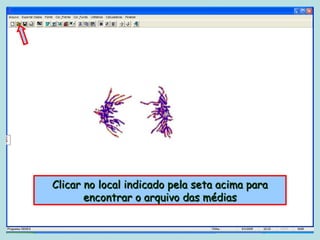
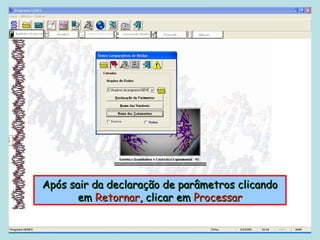
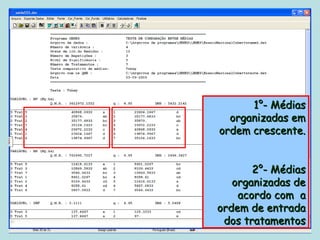
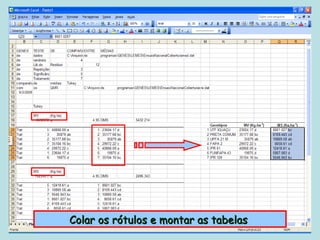
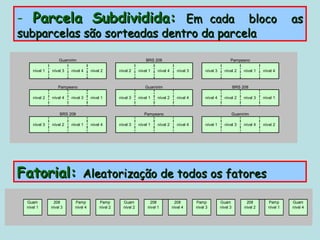
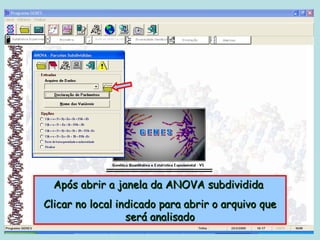
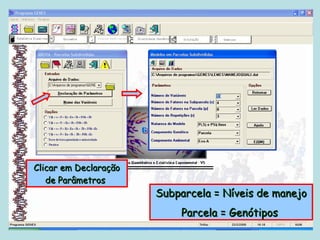
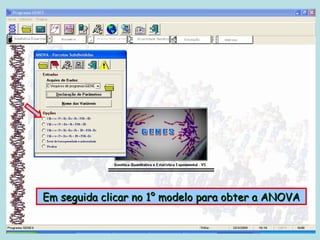
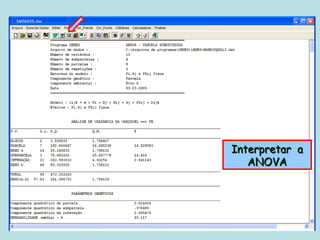
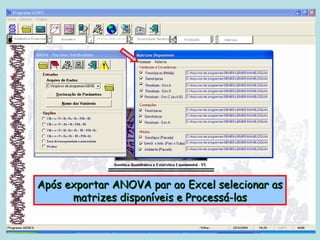
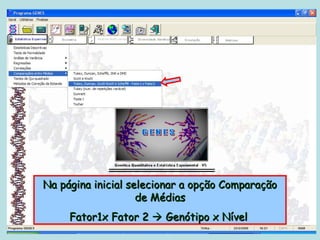
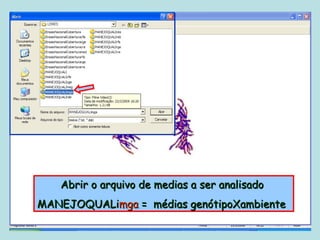
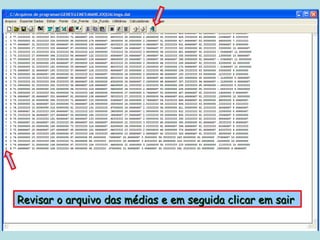
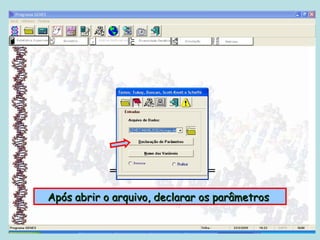
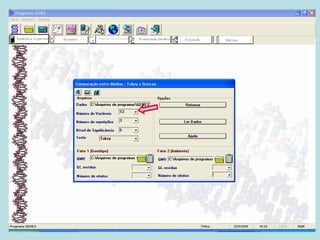
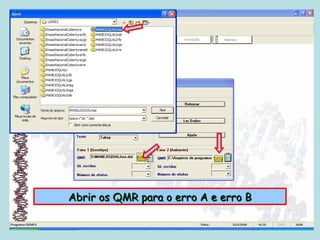
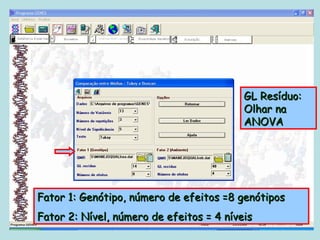
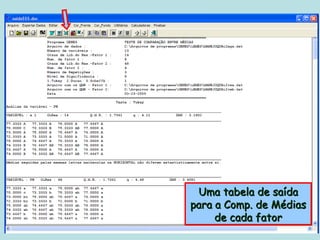
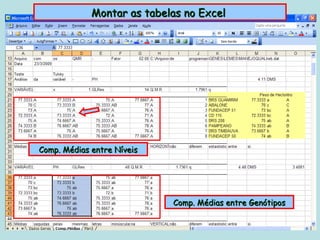
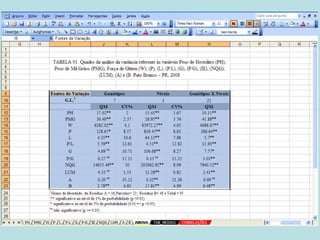
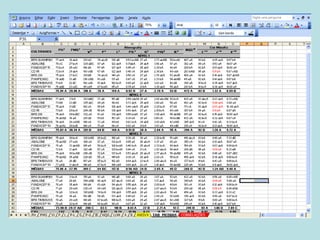
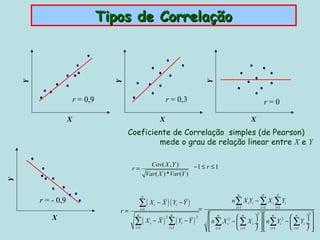
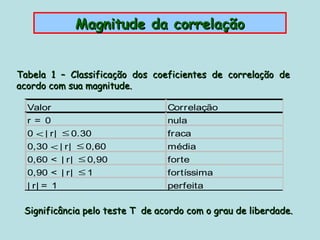
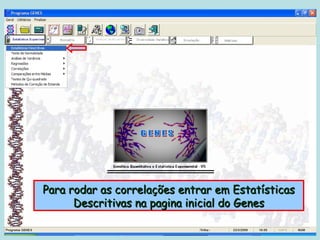
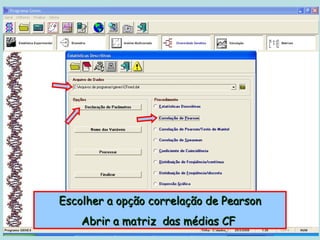
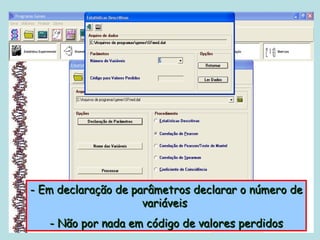
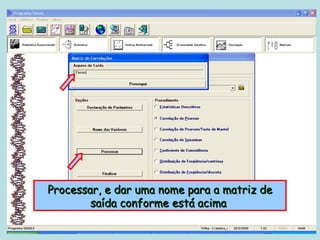
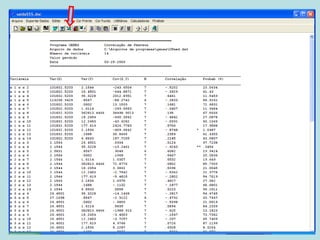
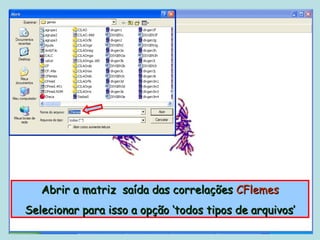
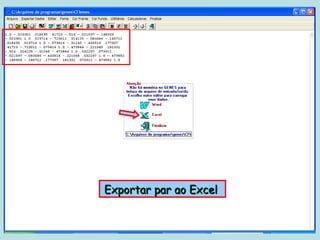
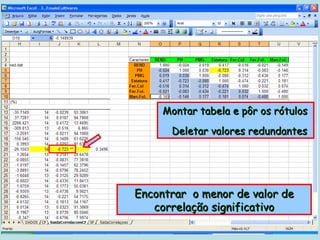
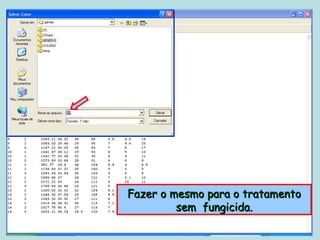
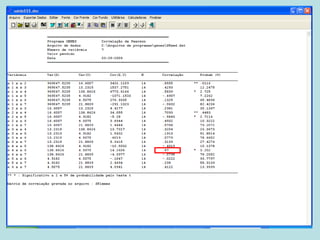
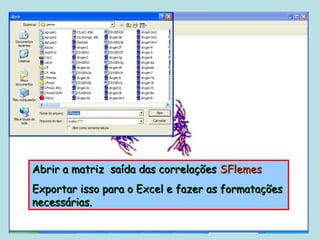
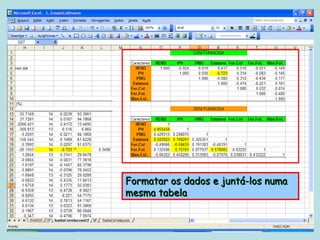
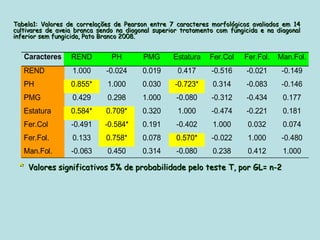
Este documento fornece instruções sobre como realizar análises estatísticas usando o programa Genes. Ele explica como carregar e salvar dados, declarar parâmetros, rodar análises de variância, comparações de médias, correlações e exportar resultados para o Excel.