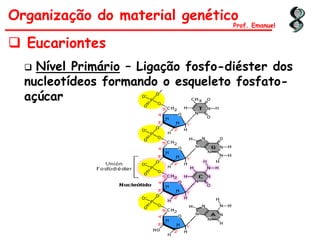
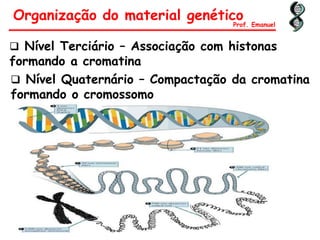
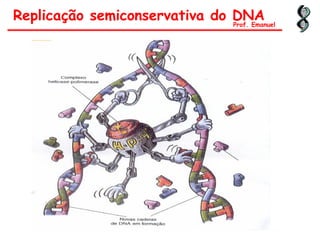
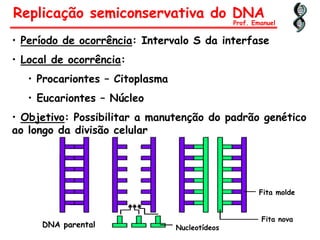
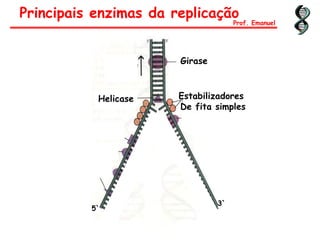
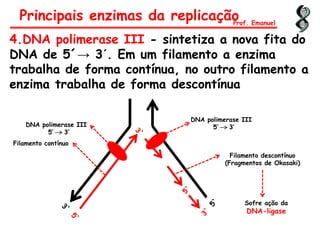
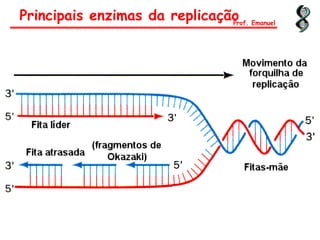
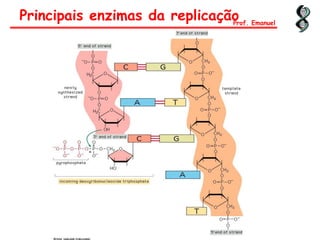
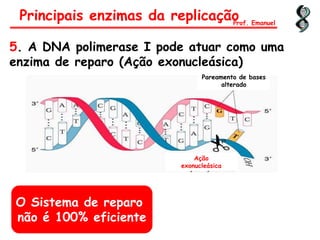
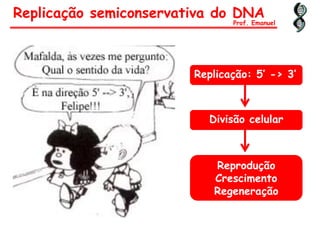
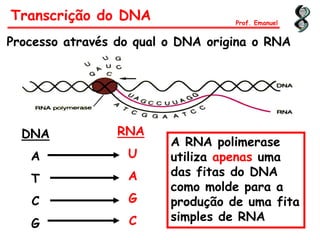
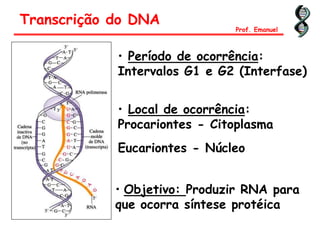
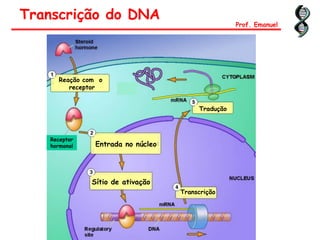
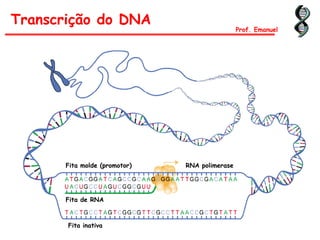
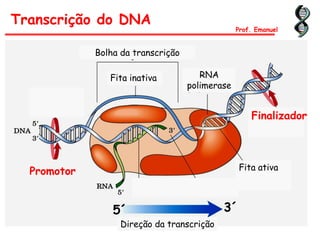
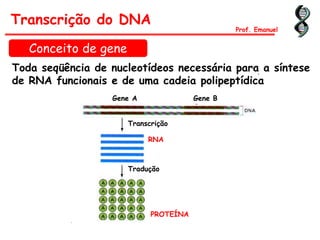
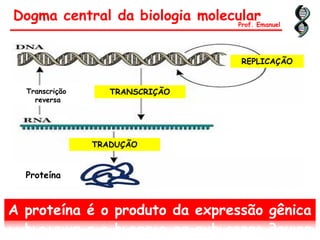
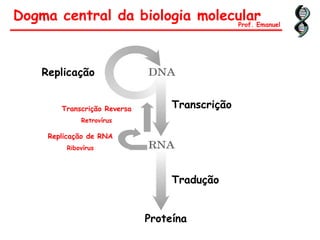
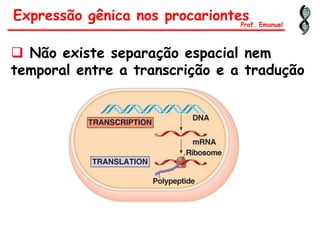
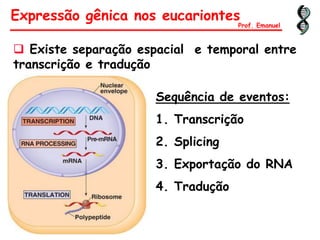
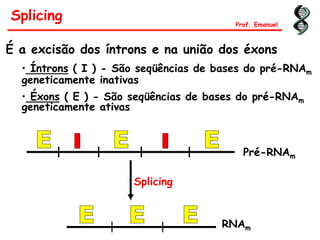
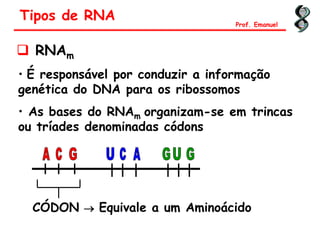
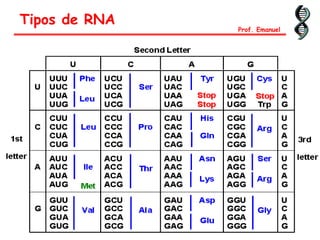
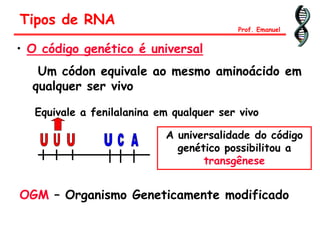
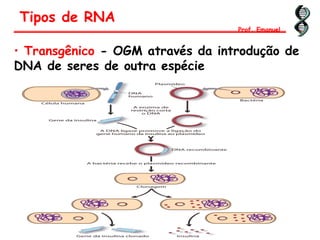
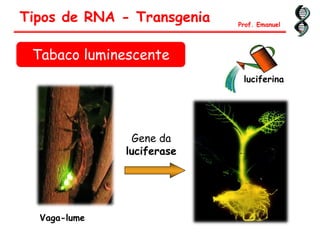
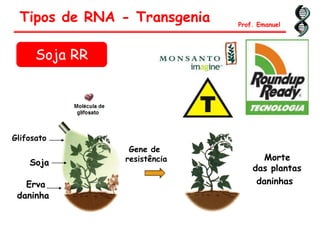
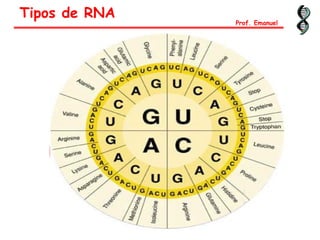
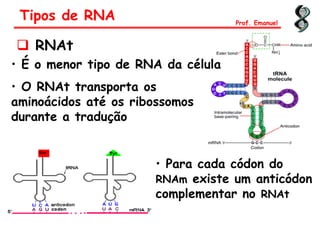
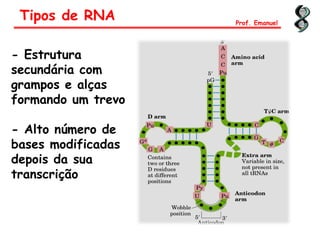
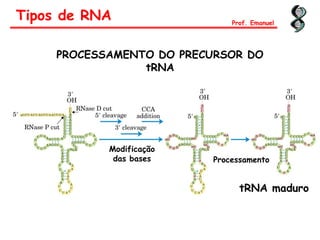
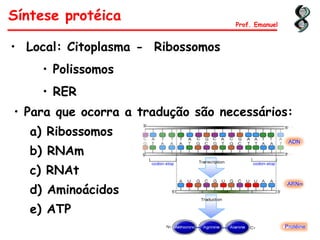
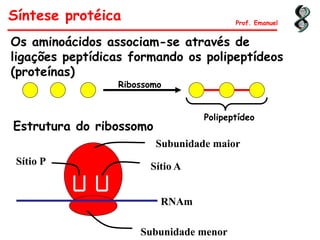
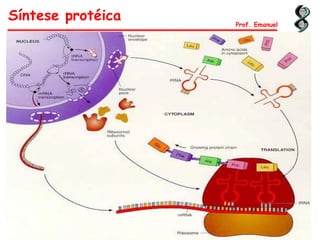
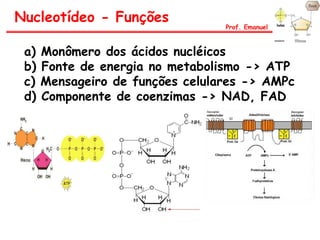
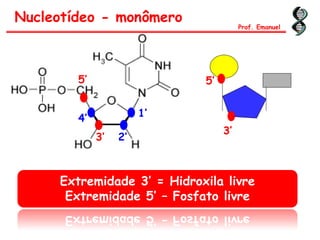
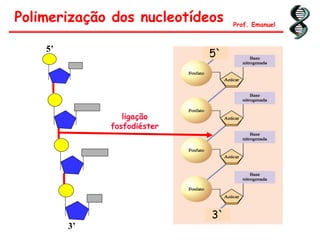
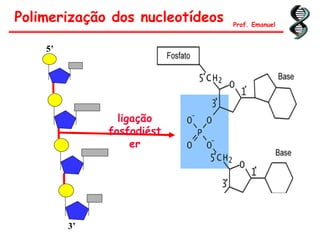
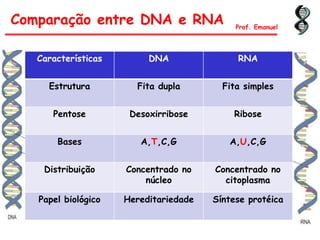
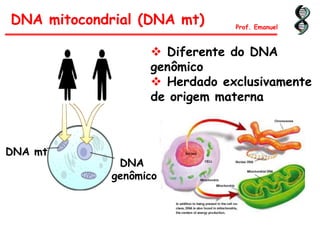
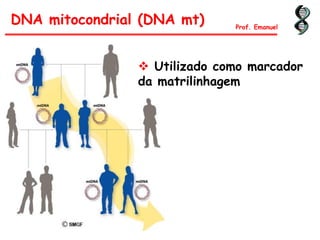
O documento discute os ácidos nucléicos, incluindo sua estrutura, composição e papel na hereditariedade e expressão gênica. Aborda os nucleotídeos, DNA, RNA, replicação, transcrição e tradução. Explica como a informação genética é copiada e expressa através destes processos para produzir proteínas.

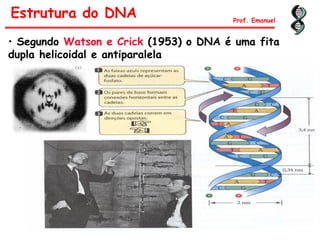


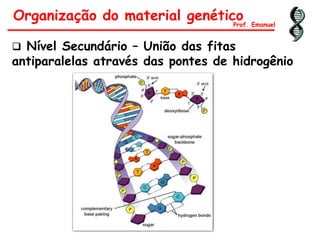
![Entre as bases nitrogenadas das
fitas antiparalelas existem pontes
de hidrogênio (estabilidade)
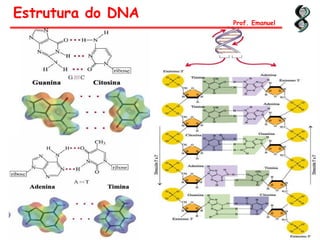
Estrutura do DNA
PAREAMENTO DAS BASES
Púrica Pirimídica
Relação de CHARGAFF
1. A + T + C + G = 100%
2. [ A ] = [ T ] e [ G ] = [ C ]
Prof. Emanuel](https://image.slidesharecdn.com/acidosnucleicos20102-130609073412-phpapp02/85/Acidos-Nucleicos-19-320.jpg)