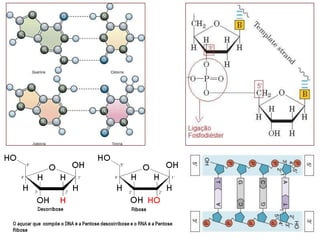
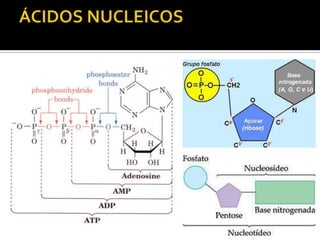
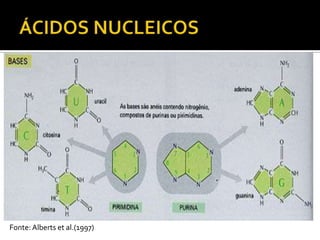
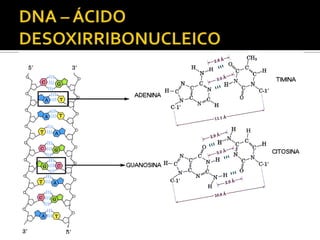
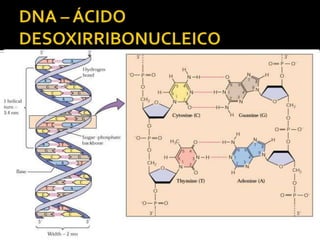
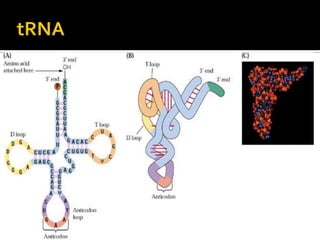
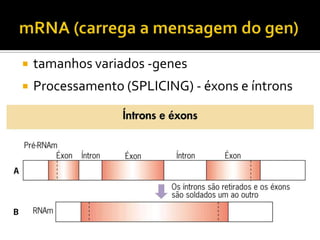
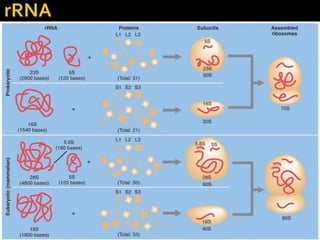
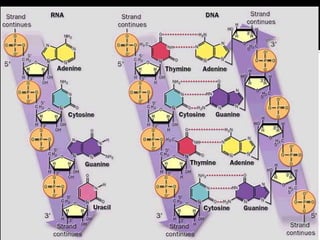
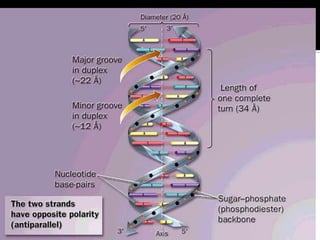
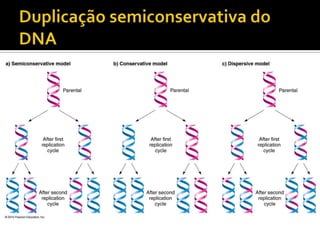
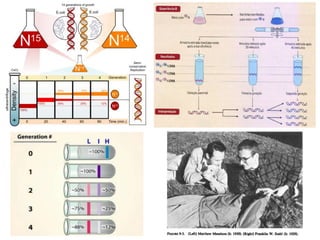
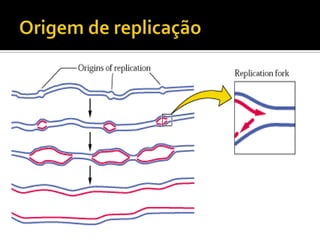
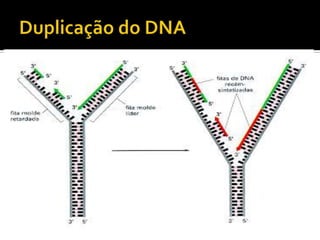
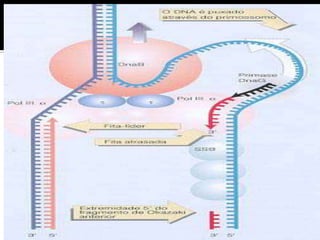
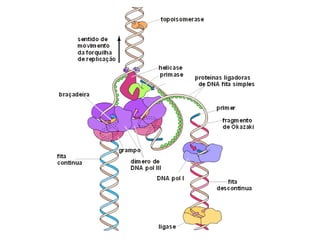
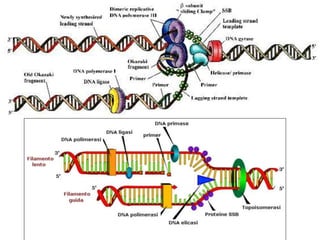
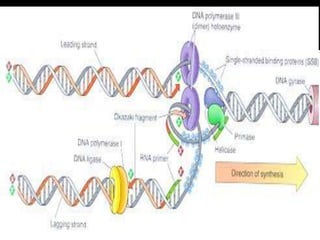
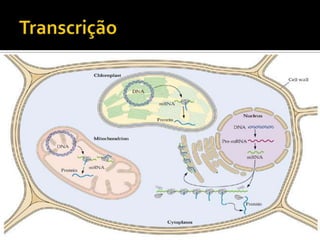
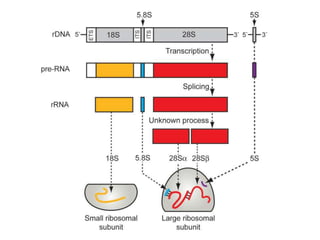
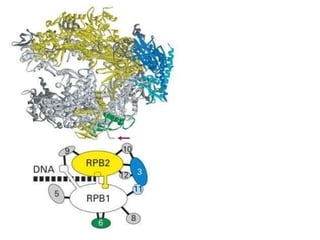
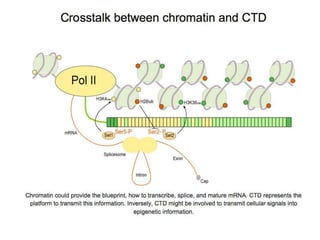
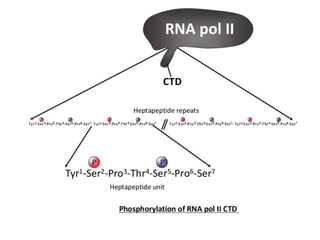
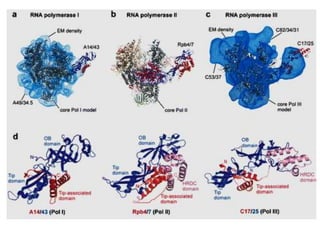
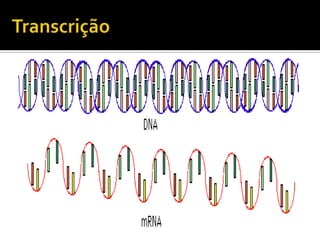
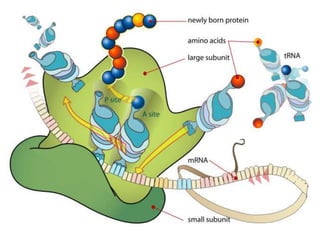
O documento descreve os principais tipos de ácidos nucleicos, DNA e RNA, e seus papéis na célula. Detalha a estrutura do DNA de fita dupla e do RNA de fita simples, assim como os processos de replicação, transcrição e tradução que usam esses ácidos nucleicos para transmitir e expressar informação genética.