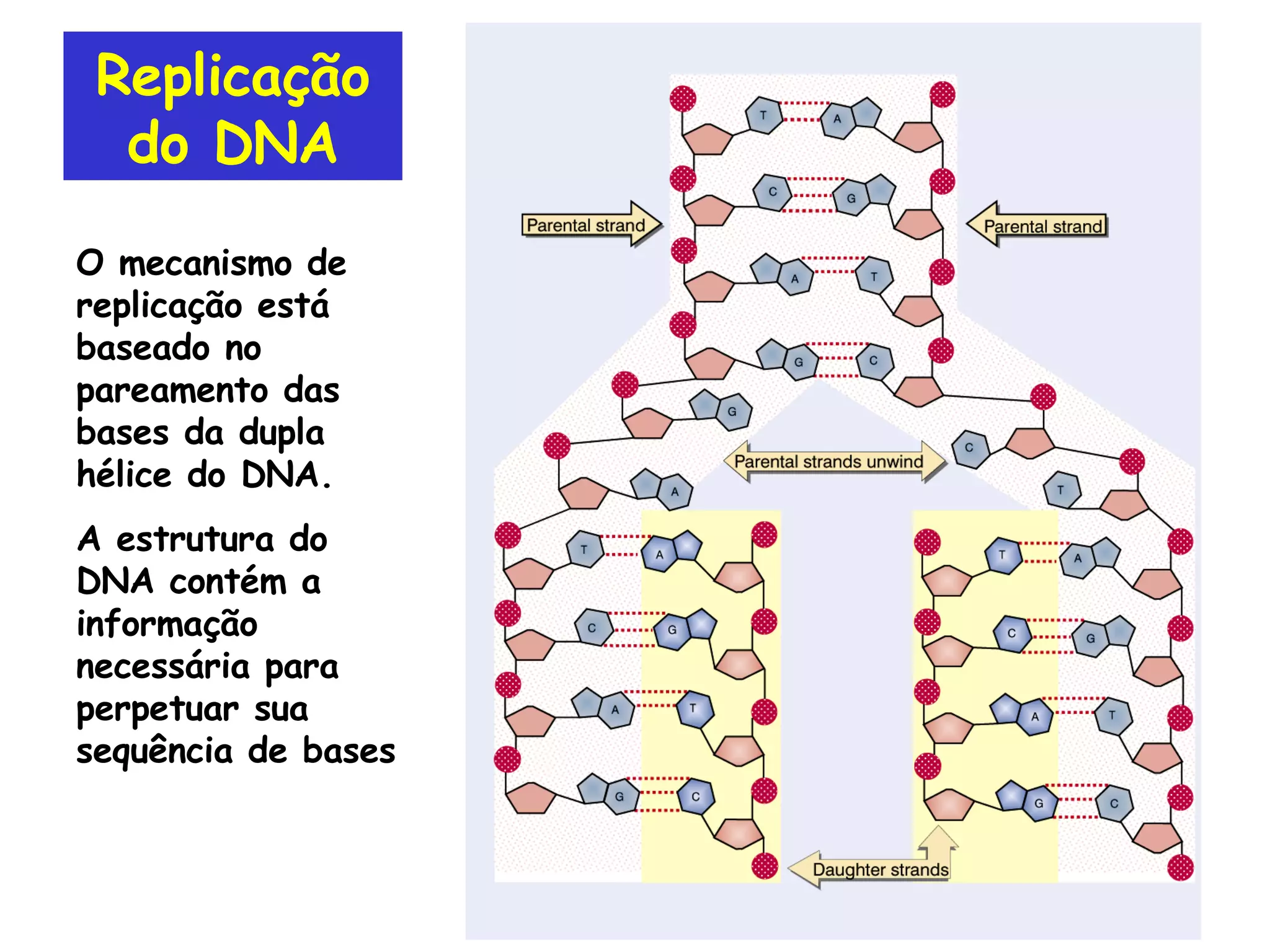
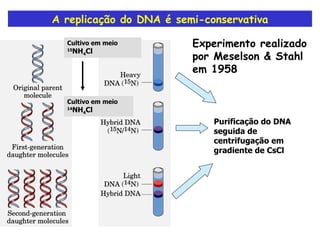
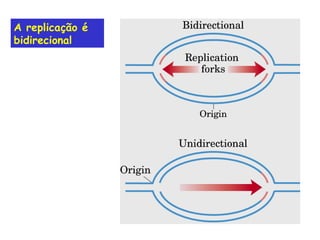
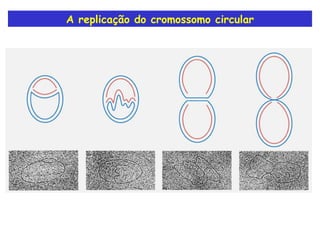
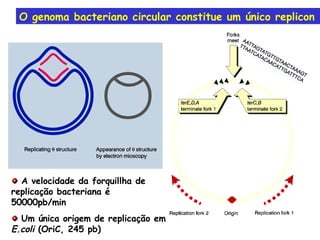
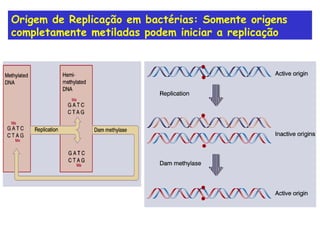
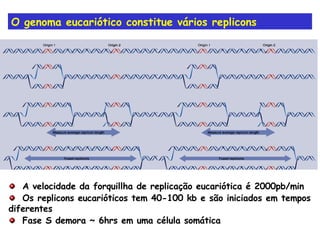
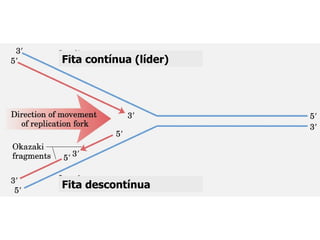
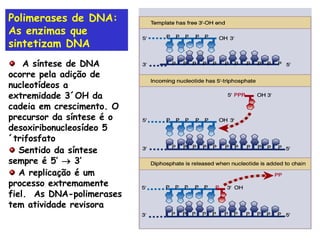
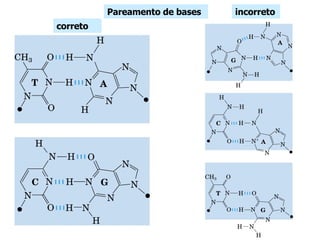
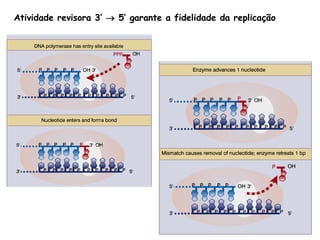
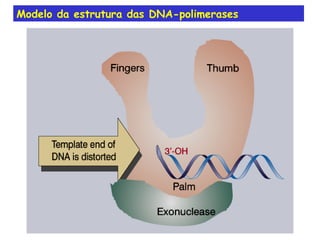
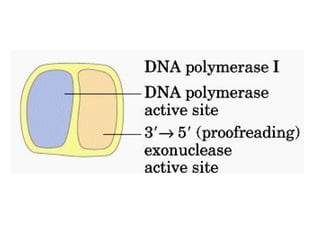
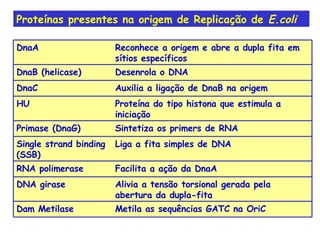
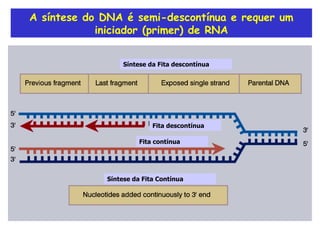
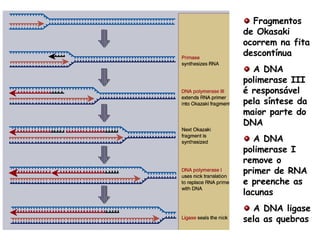
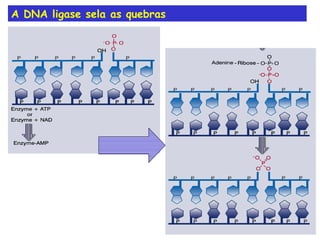
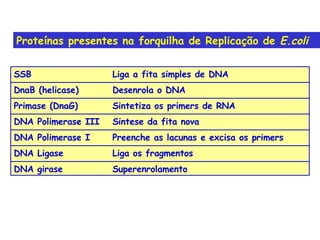
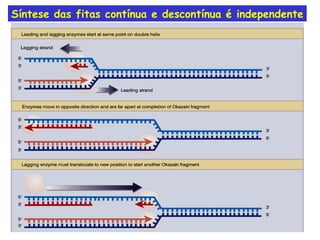
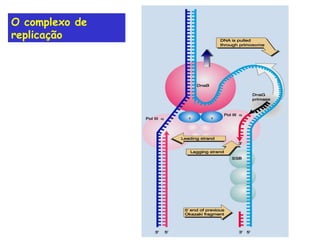
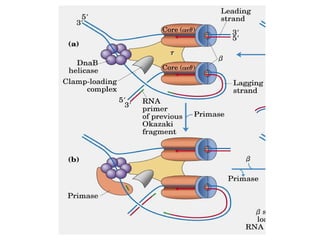
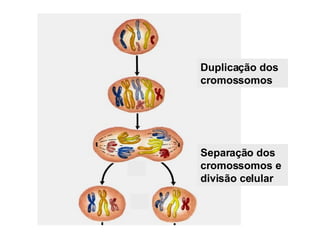
A replicação do DNA ocorre de forma semi-conservativa, com cada cadeia original servindo de molde para a síntese de uma nova cadeia complementar. A replicação é bidirecional e inicia em origens de replicação, onde proteínas reconhecem e separam a dupla hélice, permitindo a ação de helicases e primases. DNA e RNA polimerases sintetizam as novas cadeias de forma coordenada.