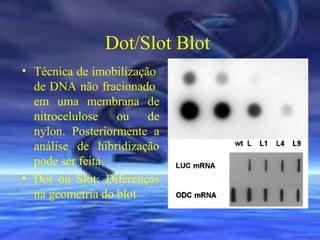
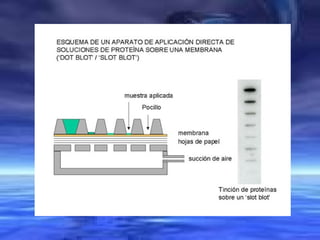
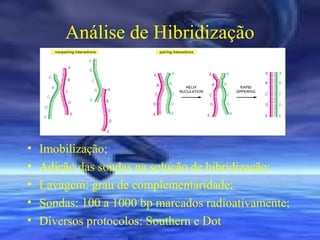
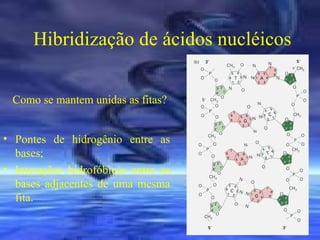
O documento descreve técnicas de análise de hibridização de ácidos nucléicos, como fatores que afetam a estabilidade dos híbridos, métodos de imobilização de DNA em membranas como Dot Blot e procedimentos para hibridização com sondas marcadas radioativamente.








![Fatores que afetam a estabilidade dos
híbridos
• Concentração de sal na solução:
∀ ↑ [sal] → ↑ estabilidade do híbrido
– Cátions monovalentes (Na+) ou
divalentes (Mg++)
– As cargas negativas dos grupos fosfato
repelem umas às outras.
– Os íons positivos da solução reduzem a
repulsão eletrostática entre as fitas.](https://image.slidesharecdn.com/analisedehibridizao-140207103722-phpapp01/85/Analise-de-hibridizacao-10-320.jpg)

![Fatores que afetam a estabilidade dos
híbridos
• pH:
↑ [OH- ]
↑ ionização dos grupos fosfato, favorecendo
a repulsão eletrostática entre as fitas.
• Concentração de formamida:
– Provavelmente forma ligações de H com os
ácidos nucléicos;
– Desestabiliza a formação de híbridos.](https://image.slidesharecdn.com/analisedehibridizao-140207103722-phpapp01/85/Analise-de-hibridizacao-12-320.jpg)

![Para DNA:DNA
Tm = 81,5 ºC + 16,6log[Na] + 41(%G+C) - 0,63(%formamida) - (500/L)
Para DNA:RNA
Tm = 79,8 ºC + 18,5log[Na] + 58,4(%G+C) + 11,8(%G+C)2 - 0,5(%formamida) - (820/L)
Para oligonucleotidos em 1 M Na+
Tm (oC) = 4 (G+C) + 2 (A+T)](https://image.slidesharecdn.com/analisedehibridizao-140207103722-phpapp01/85/Analise-de-hibridizacao-14-320.jpg)