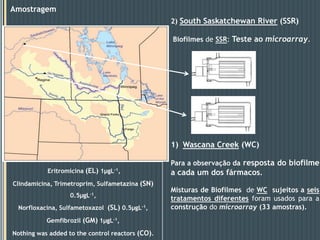
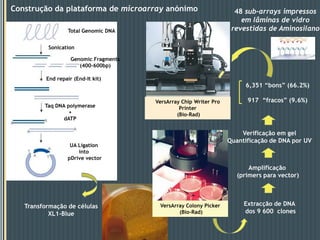
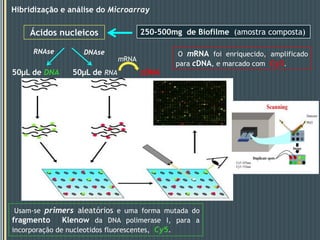
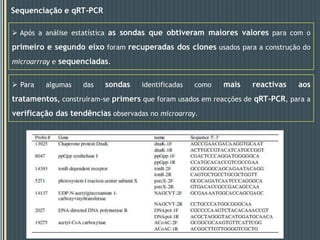
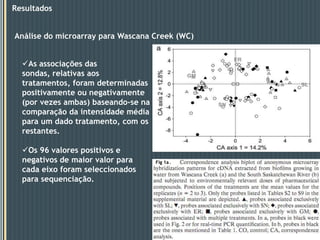
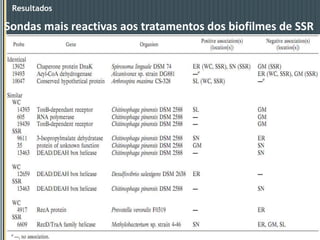
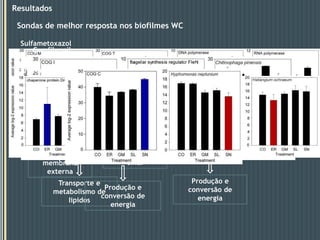
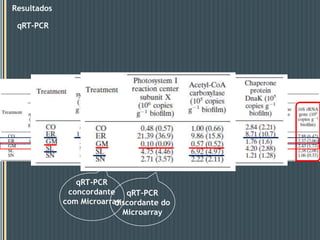
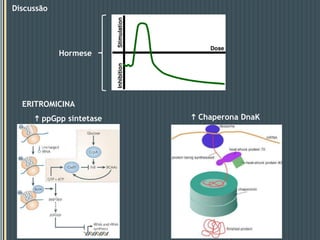
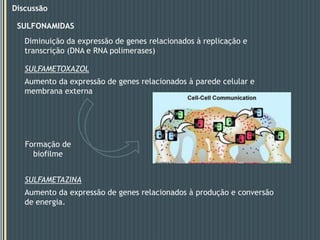
Este documento descreve um estudo que analisou a resposta de biofilmes fluviais a produtos farmacêuticos usando microarrays de DNA anônimos. Os resultados mostraram que antibióticos como eritromicina e sulfonamidas, bem como o gemfibrozil, induziram alterações na expressão gênica relacionadas a processos como produção de energia, parede celular e metabolismo lipídico. Alguns genes apresentaram respostas semelhantes em biofilmes de dois rios diferentes.