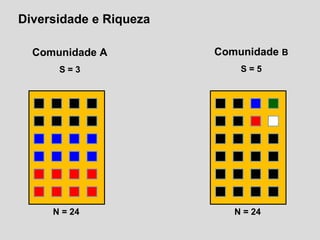
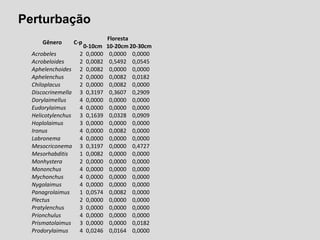
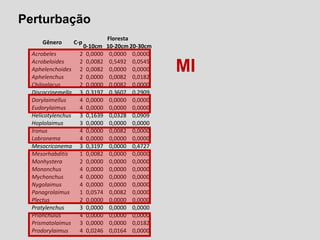
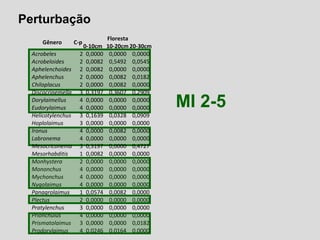
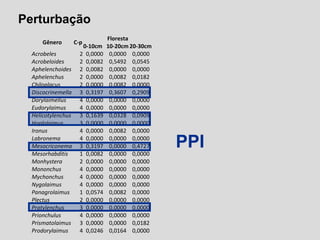
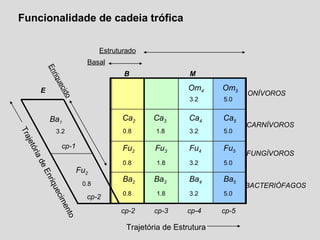
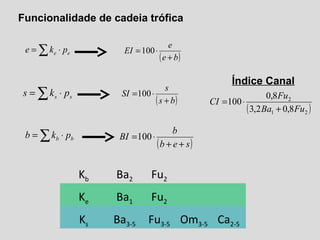
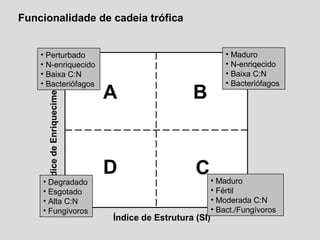
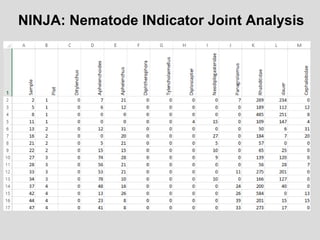
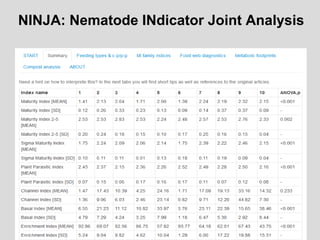
O documento aborda a utilização de nematoides para monitorar a qualidade do solo, discutindo a mensuração de comunidades de nematoides e índices ecológicos. Inclui análises estatísticas, estrutura do curso e exemplos práticos na aplicação de dados sobre diversidade e funcionalidade da cadeia trófica. Ferramentas moleculares como o sistema NINJA são mencionadas para auxiliar na análise dos dados obtidos.