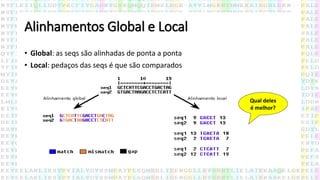
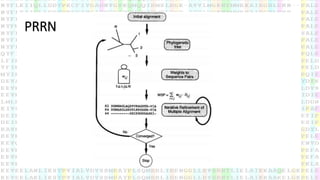
O documento discute técnicas de alinhamento múltiplo de sequências de DNA e proteínas, comparando alinhamentos globais e locais, ótimos e heurísticos. Também apresenta métodos como Dividir e Conquistar, Alinhamento Iterativo e PRRN para produzir alinhamentos múltiplos de alta qualidade de forma rápida.