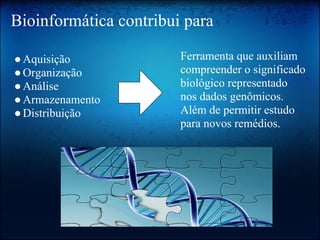
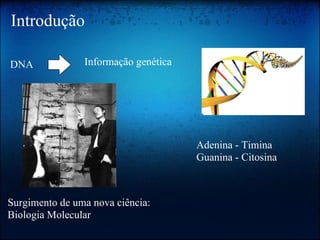
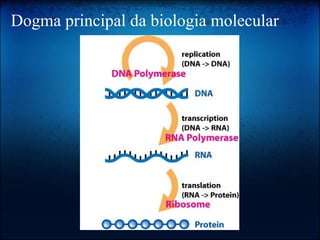
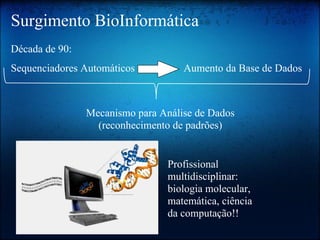
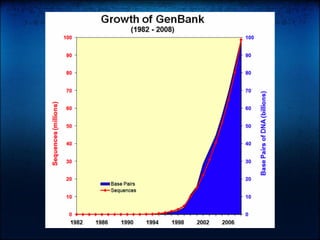
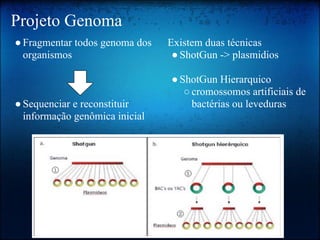
O documento discute os principais tópicos da bioinformática, incluindo seu surgimento, ferramentas como Perl e BioPerl, bancos de dados públicos como GenBank, alinhamento de sequências, o Projeto Genoma, e o uso da computação evolutiva para análises bioinformáticas.