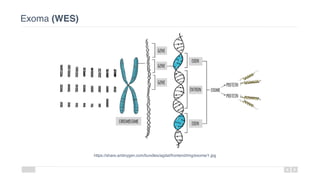
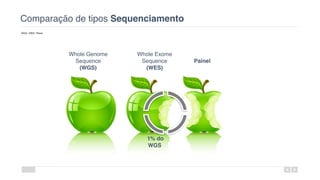
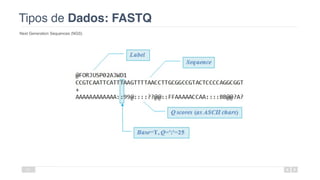
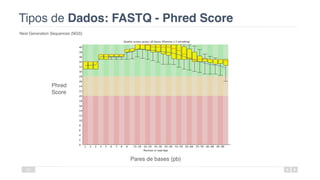
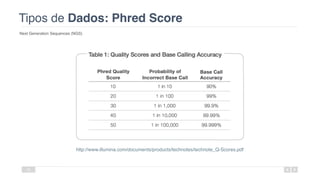
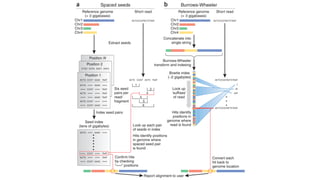
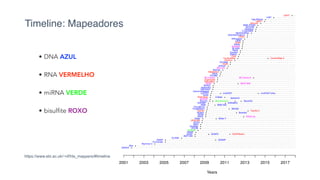
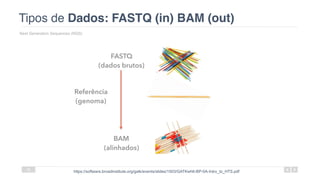
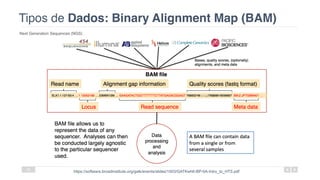
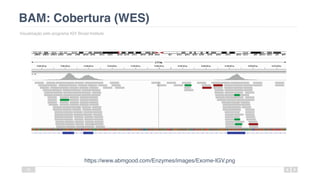
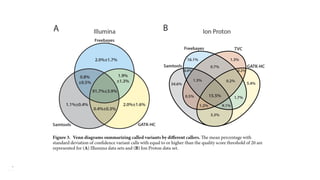
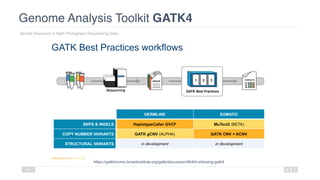
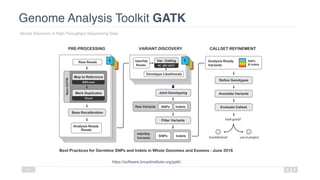
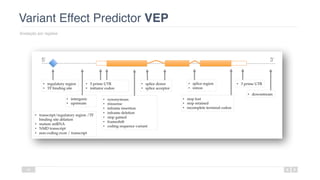
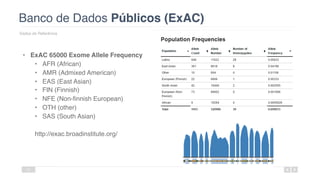
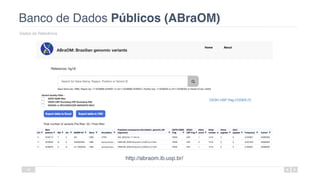
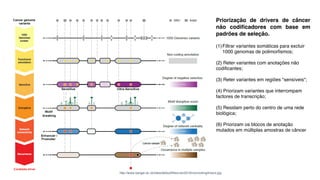
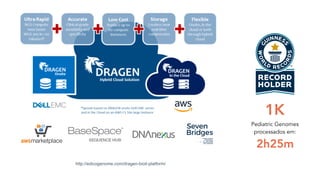
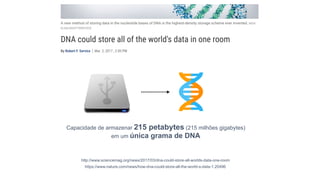
O documento aborda a bioinformática aplicada à saúde, focalizando o sequenciamento de DNA e seu impacto na medicina, incluindo o Projeto Genoma Humano e suas inovações tecnológicas. Destaca a evolução das ferramentas bioinformáticas e a importância do sequenciamento em larga escala, comparando diferentes tipos de sequenciamento como WGS e WES. Além disso, menciona recursos de dados e bancos de dados públicos para apoiar a análise genética.