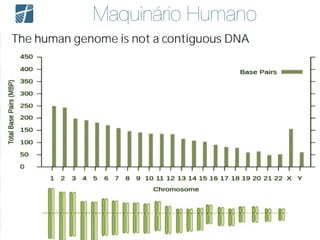
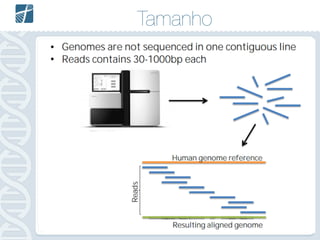
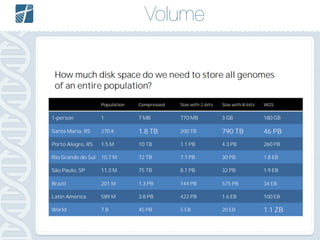
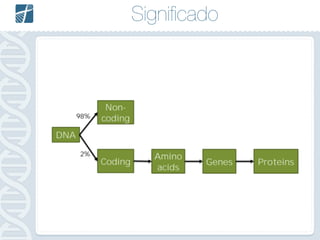
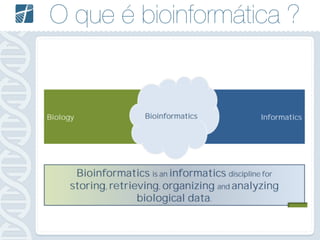
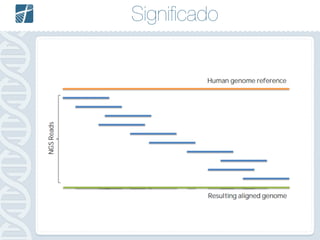
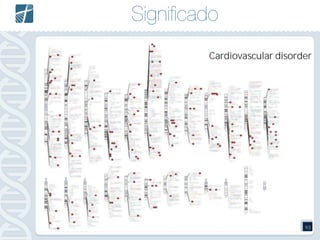
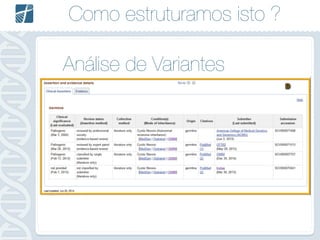
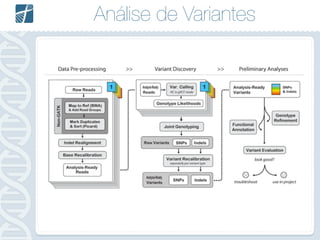
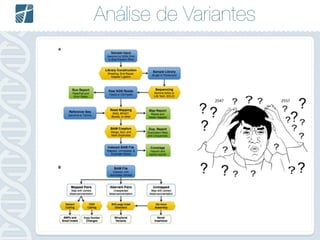
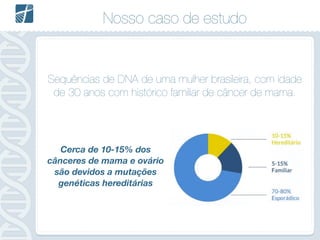
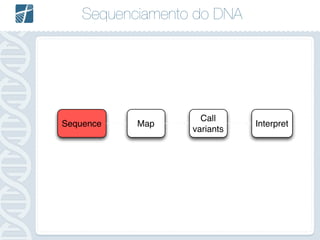
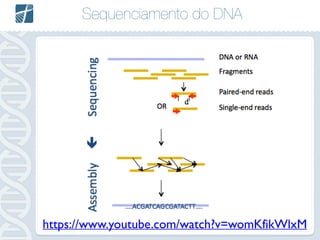
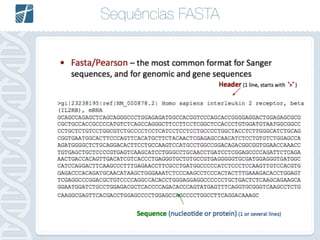
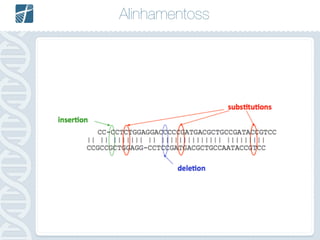
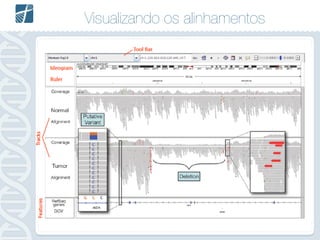
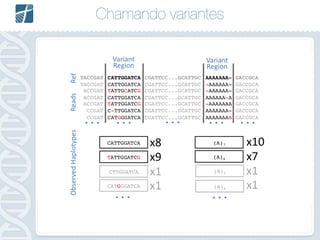
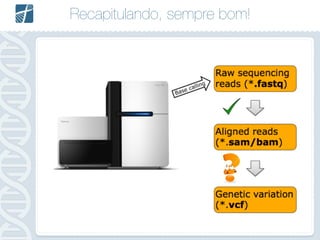
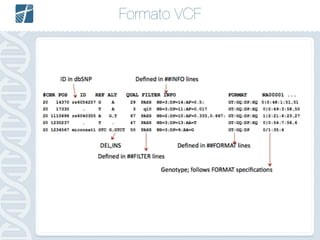
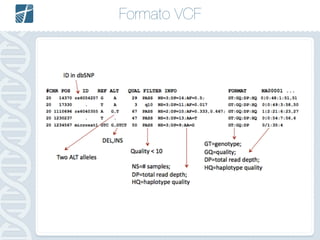
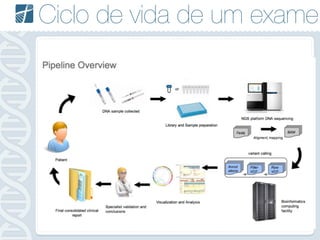
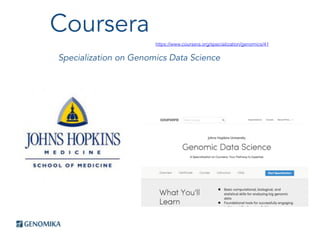
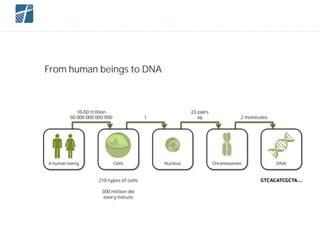
Este documento descreve como analisar seu próprio genoma usando tecnologias como Python. Apresenta os conceitos de sequenciamento de DNA, mapeamento, chamada de variantes e interpretação. Explica o fluxo de trabalho de um pipeline simples para analisar variantes em um genoma e fornece recursos para aprender mais sobre bioinformática.




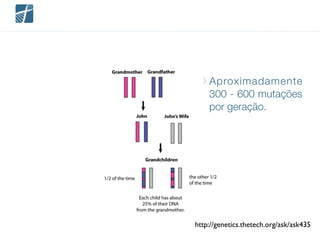




![Se quisessemos carregar o Genoma na memória ?
Como representaríamos em linguagem de programação ?
char [] humanDNA = char[ 3 200 000 000];](https://image.slidesharecdn.com/genomikasecomp2015-151024024334-lva1-app6891/85/Como-interpretar-seu-proprio-genoma-com-Python-15-320.jpg)
![Se quisessemos carregar o Genoma na memória ?
Como representaríamos em linguagem de programação ?
char [] humanDNA = char[ 3 200 000 000];](https://image.slidesharecdn.com/genomikasecomp2015-151024024334-lva1-app6891/85/Como-interpretar-seu-proprio-genoma-com-Python-16-320.jpg)
![Se quisessemos carregar o Genoma na memória ?
Como representaríamos em linguagem de programação ?
char [] humanDNA = char[ 3 200 000 000];](https://image.slidesharecdn.com/genomikasecomp2015-151024024334-lva1-app6891/85/Como-interpretar-seu-proprio-genoma-com-Python-17-320.jpg)