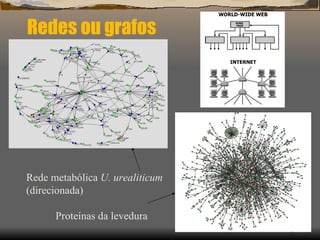
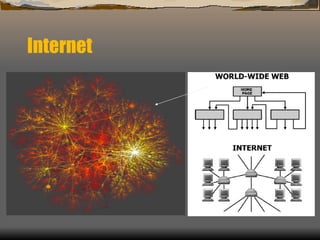
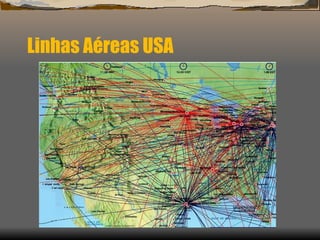
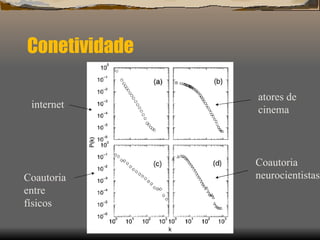
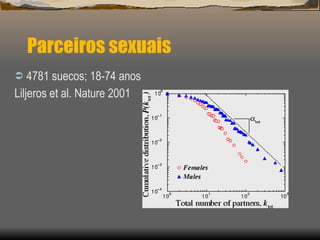
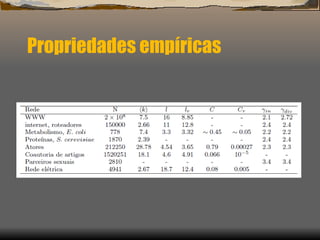
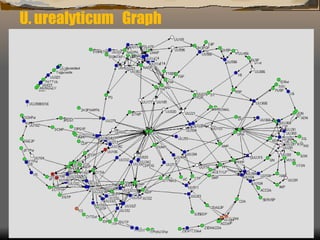
1) O documento discute as propriedades de redes complexas como redes metabólicas, a internet e redes de interação social.
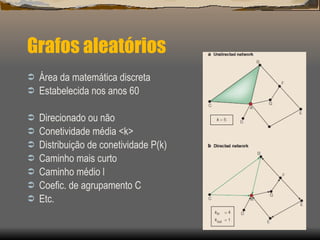
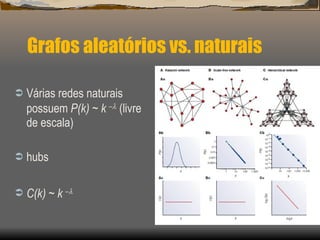
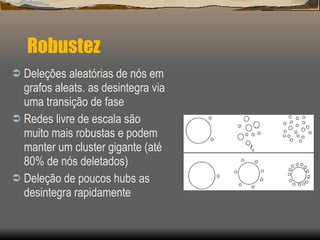
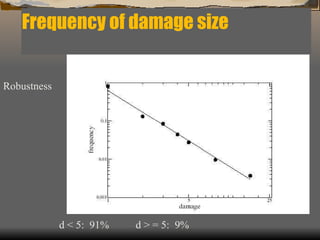
2) Apresenta modelos matemáticos como o modelo de Barabási-Albert para gerar redes livres de escala e discute a robustez dessas redes.
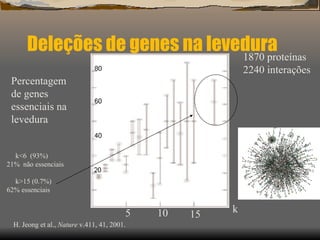
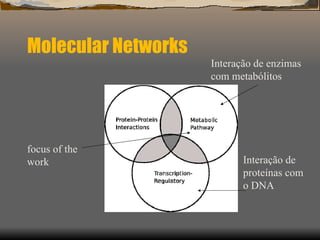
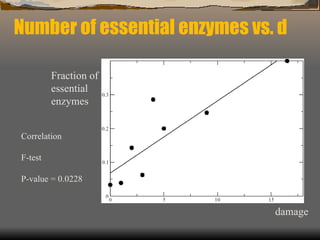
3) Discutem dois estudos: prever a essencialidade de enzimas em redes metabólicas e incluir a atividade gênica em redes moleculares.









![Referências para leitura Barabási, Reviews of Modern Physics (2002) Barabási, Nature Reviews (2004) Mombach, CNMAC (2004) Livro Edição especial, Science (2009) http://icensa.nd.edu/books.html email: [email_address] Mostrar outros livros](https://image.slidesharecdn.com/mombach-101229070315-phpapp02/85/A-ciencia-das-redes-complexas-20-320.jpg)














![Referências para leitura Barabási, Reviews of Modern Physics (2002) Barabási, Nature Reviews (2004) Mombach, CNMAC (2004) Livro Edição especial, Science (2009) email: [email_address]](https://image.slidesharecdn.com/mombach-101229070315-phpapp02/85/A-ciencia-das-redes-complexas-41-320.jpg)
