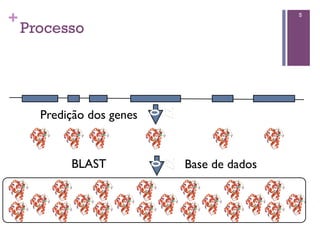
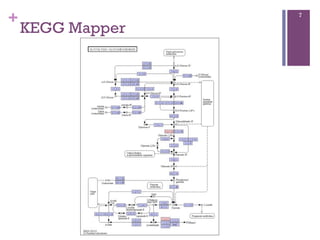
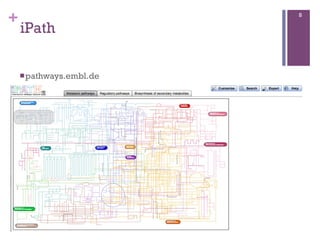
Este documento discute análise funcional de bioinformática. Ele descreve ferramentas como BLAST e HMMER para alinhar sequências e bancos de dados como COG, KEGG e PFam para anotar genes. Também fornece dicas para escolher os melhores hits e cobrir múltiplas bases de dados para melhor caracterizar proteínas e mapear vias metabólicas.