1) TASSEL é um software para mapeamento por associação de traços complexos usando marcadores genéticos como SNPs.
2) Ele permite identificar associações entre o fenótipo e marcadores genéticos através de três pipelines: Discovery, Production e UNEAK.
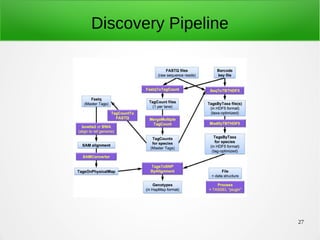
3) O pipeline Discovery usa um genoma de referência para mapear QTLs em vários passos executados por plugins, enquanto o Production usa informações do Discovery em um único passo.

![2
Mapeamento por associação
● Software para o mapeamento por associação
de traços (características) complexos [1].](https://image.slidesharecdn.com/tassel-150619161430-lva1-app6892/85/TASSEL-Trait-Analysis-by-aSSociation-Evolution-and-Linkage-2-320.jpg)


![5
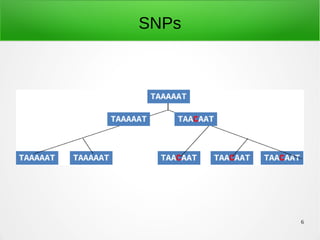
SNPs
● Single Nucleotide Polymosphisms
● Grande abundância
– A cada 300 ~ 600 nucleotídeos [2]
● Tecnologias de genotipagem
● Mutações que se propagaram ao longo de
gerações](https://image.slidesharecdn.com/tassel-150619161430-lva1-app6892/85/TASSEL-Trait-Analysis-by-aSSociation-Evolution-and-Linkage-5-320.jpg)



















![31
Referências
● [1]
http://bioinformatics.oxfordjournals.org/content/
23/19/2633.full.pdf
● [2]
http://www.lge.ibi.unicamp.br/lgeextensao2008/
extsup/snps.pdf
● Wiki Tassel:
– https://bitbucket.org/tasseladmin/tassel-5-source/wi
ki/Home](https://image.slidesharecdn.com/tassel-150619161430-lva1-app6892/85/TASSEL-Trait-Analysis-by-aSSociation-Evolution-and-Linkage-31-320.jpg)